# install.packages("pacman")
library(pacman)
p_load_gh('jorvlan/raincloudplots') # レインクラウドプロット
p_load(tidyverse, # データセットのハンドリング
causaldata, # データセット集
naniar, # 欠測値の処理・可視化
visdat, # データ構造の可視化
ggbeeswarm, # ビー・スウォームプロット
GGally # 多変量の可視化
)
data(nhefs) # データの呼び出しPart 2: 記述統計の可視化
1 Package
2 Data
本パートでは、causaldata packageに格納されているNational Health and Nutrition Examination Survey Data I Epidemiologic Follow-up Study (NHEFS) の nhefs データセットを用います。このデータセットは、“Causal Inference: What If” by Hernán and Robinsでも具体例として使用されています。
まずは、nhefs データセットは何人のデータで、いくつの変数があるか、dim() で確認します。
dim(nhefs)[1] 1629 67[1] 1629 67 とあるように、1629行(人数)で67列(変数)があることが分かります。
簡単にデータセットを確認したい時には、head() で確認できます。
head(nhefs)# A tibble: 6 × 67
seqn qsmk death yrdth modth dadth sbp dbp sex age race income
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <fct> <dbl> <fct> <dbl>
1 233 0 0 NA NA NA 175 96 0 42 1 19
2 235 0 0 NA NA NA 123 80 0 36 0 18
3 244 0 0 NA NA NA 115 75 1 56 1 15
4 245 0 1 85 2 14 148 78 0 68 1 15
5 252 0 0 NA NA NA 118 77 0 40 0 18
6 257 0 0 NA NA NA 141 83 1 43 1 11
# … with 55 more variables: marital <dbl>, school <dbl>, education <fct>,
# ht <dbl>, wt71 <dbl>, wt82 <dbl>, wt82_71 <dbl>, birthplace <dbl>,
# smokeintensity <dbl>, smkintensity82_71 <dbl>, smokeyrs <dbl>,
# asthma <dbl>, bronch <dbl>, tb <dbl>, hf <dbl>, hbp <dbl>,
# pepticulcer <dbl>, colitis <dbl>, hepatitis <dbl>, chroniccough <dbl>,
# hayfever <dbl>, diabetes <dbl>, polio <dbl>, tumor <dbl>,
# nervousbreak <dbl>, alcoholpy <dbl>, alcoholfreq <dbl>, …今回は変数が多いので、全部が表示しきれません。他の列も確認したい場合は、View(nhefs) でエクセルのように、データを確認することができます。また、コマンドを使わずに、Environmentタブ内の当該データ名をクリックしても確認できます。
Part 3でも使用する累積的な喫煙の指標 Pack-Year の計算を行って、新しい列(pack_year_n)を mutate() で作成したいと思います。
データのハンドリングについては、こちらを参照ください。
nhefs01 <- nhefs |>
mutate(pack_years_n = (smokeintensity / 20) * smokeyrs)もう一度、データセットの列数を確認してみると、pack_years_nの列が追加され、68列になっていることが分かります。
dim(nhefs01)[1] 1629 683 Overview & Missing values(欠測値)
まずは、データの概観を可視化してみます。どんなタイプの変数が多いのか、NA はどの程度あるのか、確認します。
本セミナーでは、nhfes データ全体を使用していますが、実践的には解析に使用する変数に絞って描画すると、変数名もはっきりと認識できるグラフを作成できます。
3.1 Overview
ここでは、visdat パッケージの vis_dat() を使用してデータセットにどのようなデータ型1の変数が含まれているか確認しています2。
vis_dat(nhefs01) 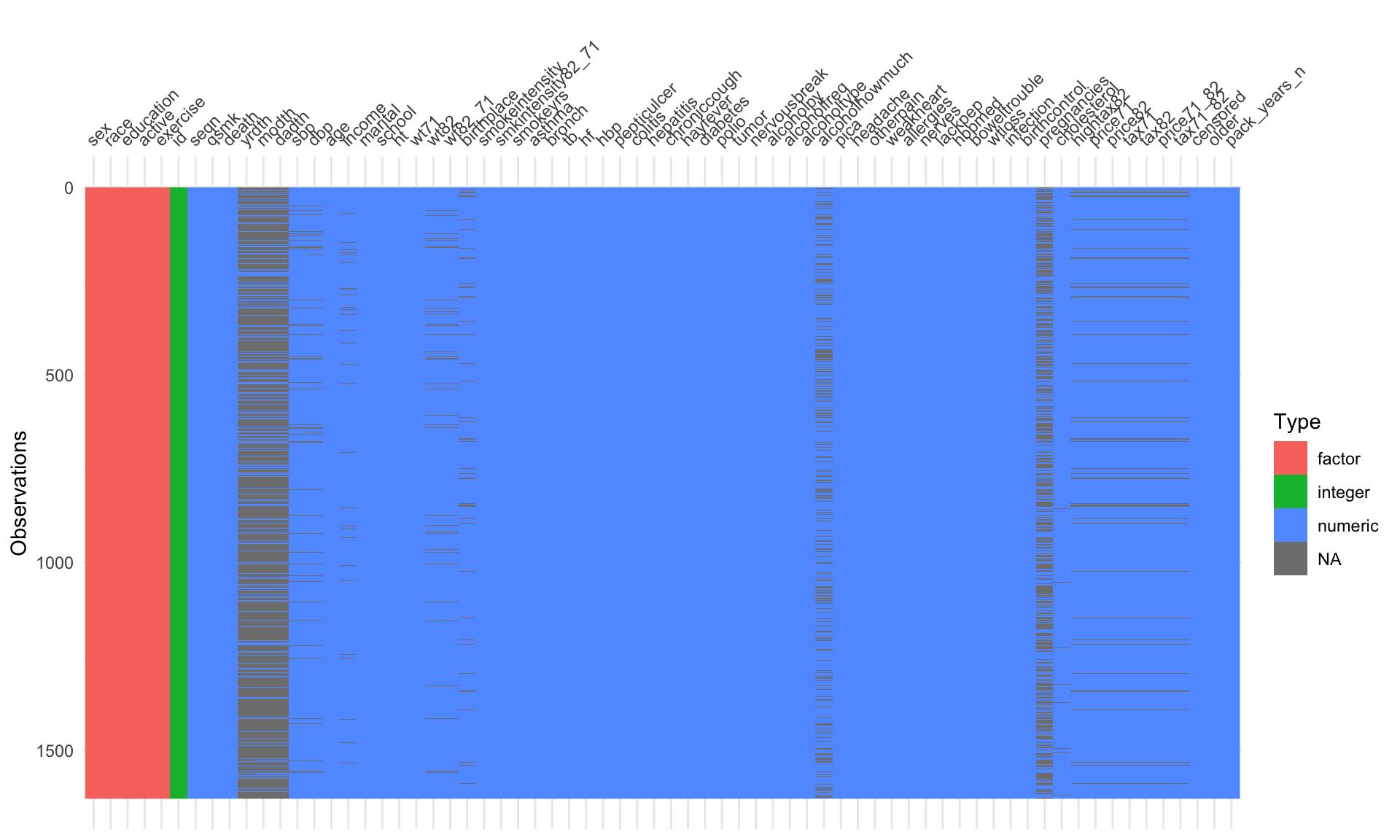
3.2 Missingness at each variable
今度は、naniar パッケージのgg_miss_var() を使用して欠測値の可視化を行ってみます3。
この関数では、各変数でどの程度(割合)欠測値 NA があるかをロリーポッププロット(点と線)で表示しています。
gg_miss_var(nhefs01, show_pct = TRUE)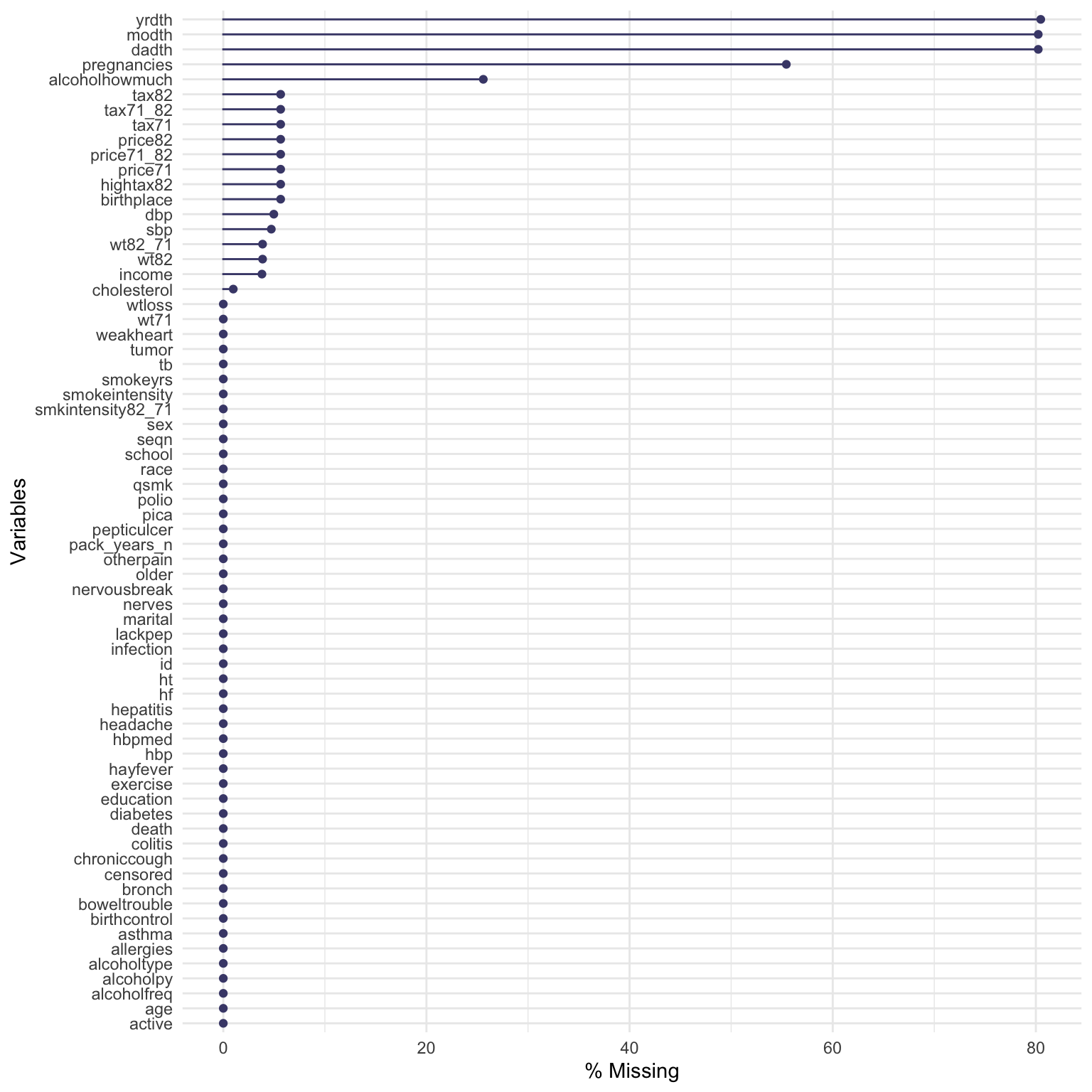
数値で欠測値の割合を確認したい場合には、miss_var_summary() を使用します。なお、order 引数に TRUE を入れることで多い順番に並び替えることができます。
n_miss 列には絶対数が、pct_miss 列には割合が表示されています。
miss_var_summary(nhefs01, order = TRUE)# A tibble: 68 × 3
variable n_miss pct_miss
<chr> <int> <dbl>
1 yrdth 1311 80.5
2 modth 1307 80.2
3 dadth 1307 80.2
4 pregnancies 903 55.4
5 alcoholhowmuch 417 25.6
6 birthplace 92 5.65
7 hightax82 92 5.65
8 price71 92 5.65
9 price82 92 5.65
10 tax71 92 5.65
# … with 58 more rows3.3 Missingness relationship between variables
ここまでは、ある一つの変数についての確認でしたが、解析で大きく問題になるのは、NAが変数間で独立していない場合(NA の発生状況が変数間で似ている)です。
そこで、二変数やデータ全体での欠測値の発生状況についても確認する必要があります。
3.3.1 全ての変数
まずは、データ全体での NA の共有状況を vis_dat パッケージの vis_miss() を使用して可視化します。今回は、cluster 引数を TRUE にしているので、NA の共有状況をある程度把握することができます。
vis_miss(nhefs01, cluster = TRUE)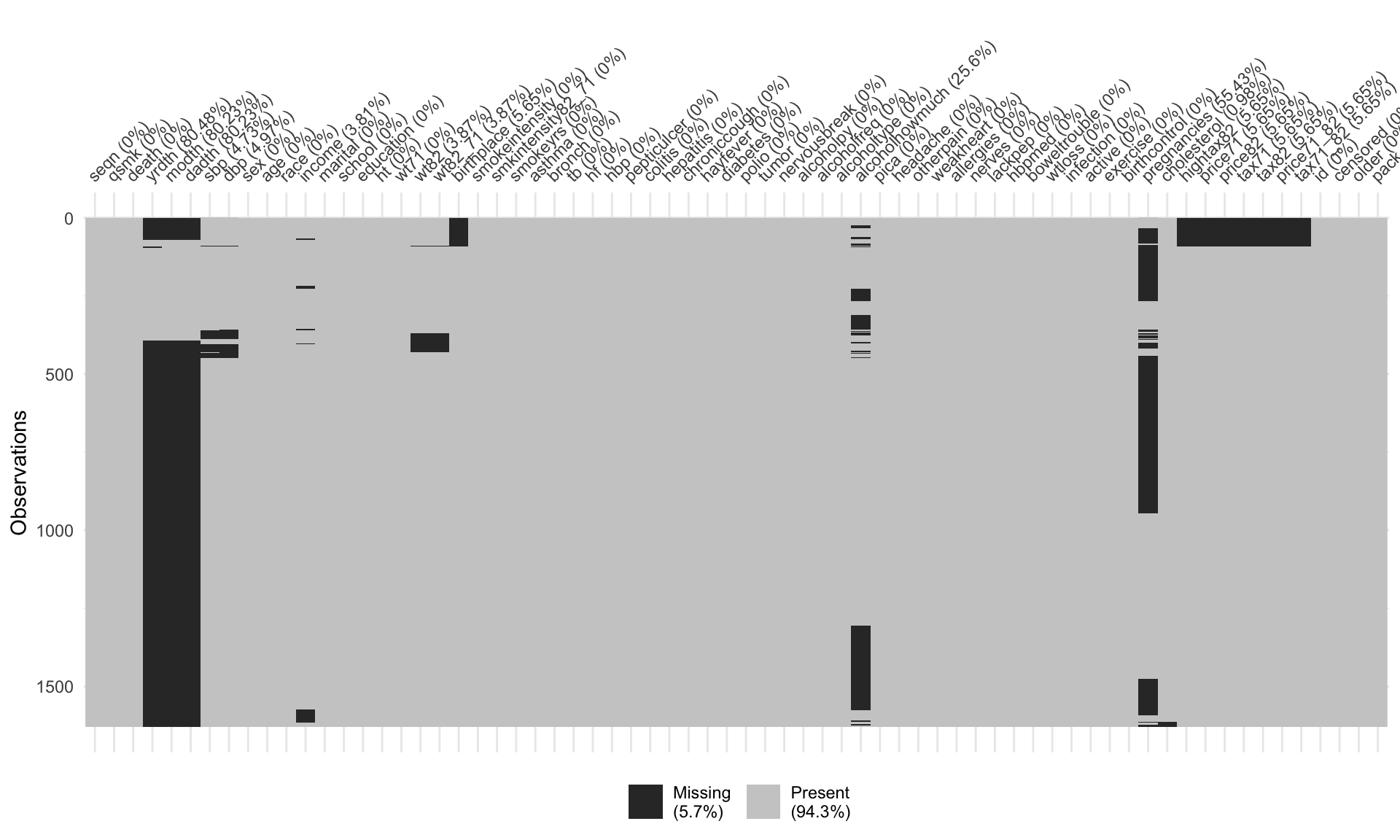
3.3.2 連続変数 × 名義変数
次に、死亡の有無(death)で層別化した場合に、特定の変数(コレステロール)のNAの割合に違いがあるかを確認します。
dplyr パッケージの group_by() によって層別化した後に、先ほど使用した miss_var_summary() で欠測値の確認、さらに filter() によって変数を指定しています。
nhefs01 |>
# データの層別化
group_by(death) |>
# 欠測値の割合の確認
miss_var_summary() |>
# cholesterolのみを指定
filter(variable == "cholesterol")# A tibble: 2 × 4
# Groups: death [2]
death variable n_miss pct_miss
<dbl> <chr> <int> <dbl>
1 0 cholesterol 16 1.22
2 1 cholesterol 0 0 3.3.3 連続変数 × 連続変数
最後に、2つの連続変数間について、一方が NA の場合にもう一方の変数の分布がどのようになっているかを確認します。
naniar パッケージの geom_miss_point() を使用しています。青色がどちらの変数も観測されている場合、赤色がどちらかの変数に NA がある場合を示しています。
ggplot(nhefs01, aes(x = pack_years_n, y = cholesterol)) +
# 欠測値の描画
geom_miss_point(size = 3, alpha = 0.6) +
# 背景の設定
theme_bw() +
# 軸や凡例の文字サイズの設定
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 20),
legend.title = element_text(size = 20),
legend.text = element_text(size = 18),
strip.text.x = element_text(size = 18))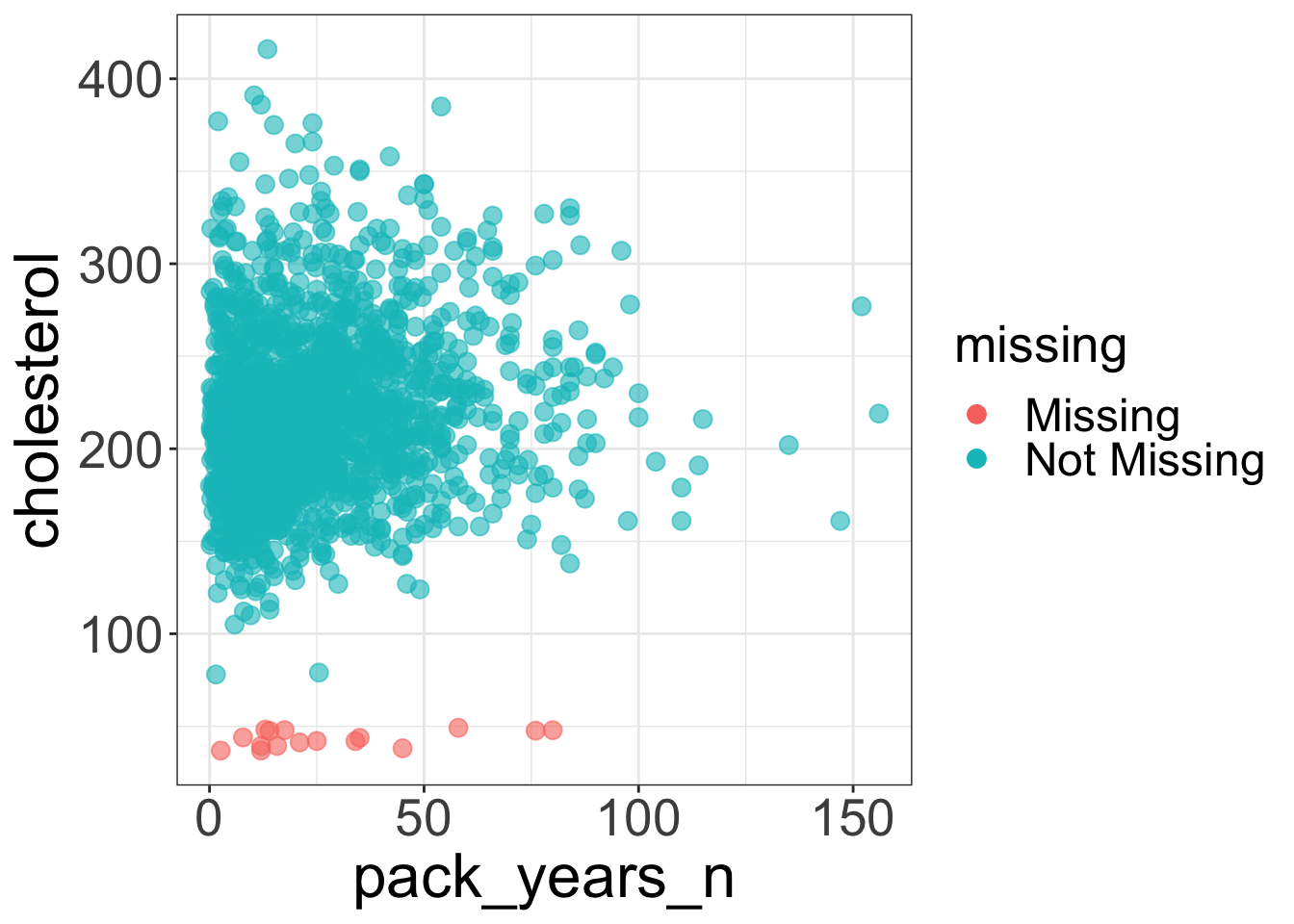
散布図の下部にある赤色のポイントは cholesterol が欠測している人の pack_years_n の分布を示し、(今回は観察されていないが)分布の左にある赤色のポイントがあればpack_years_n が欠測している人の cholesterol の分布を示します。
今回の図の場合は、「cholesterol に欠測が発生している人でも、pack_years_n の分布は、cholesterol が観察されている人のそれと概ね同じである」という解釈です。
4 Continuous variable (連続変数)
まずは、Part 3でも使用する Pack-Year を用いて、連続変数(一変量および二変量)の可視化方法を確認していきます。
4.1 ヒストグラム
ヒストグラムには、geom_histogram() を使用します。今回は一変量なので、aes() 内は、x に pack_years_n を指定しています。
ggplot(nhefs01, aes(x = pack_years_n)) +
# ヒストグラムの描画(枠線を黒に指定)
geom_histogram(color = "Black") +
theme_bw() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 20)) +
labs(x = "Pack-Year", y = "Count")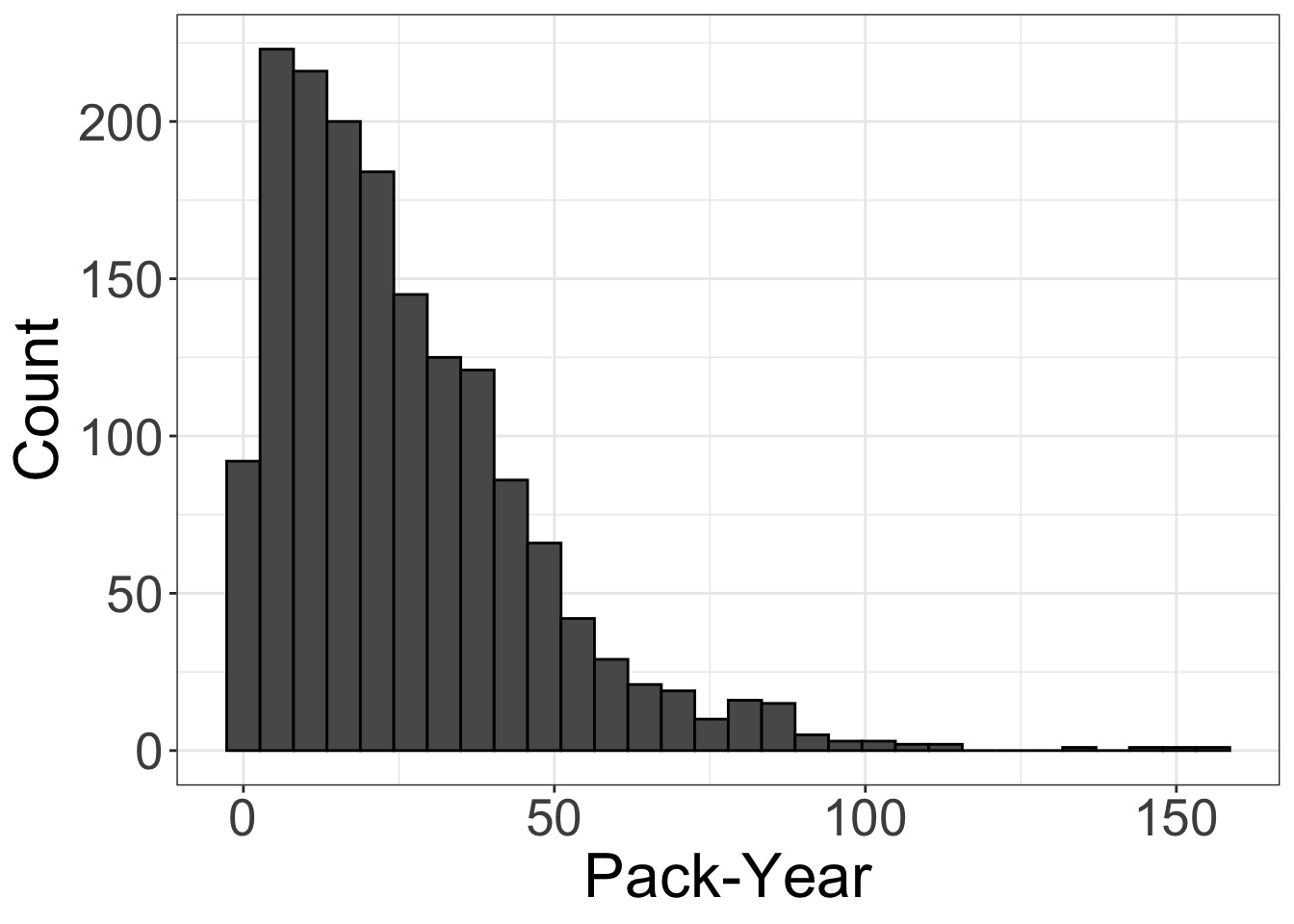
4.2 密度関数プロット
密度関数プロットには、geom_density() を使用します。今回は死亡の有無(death)を層別化に使っているので、aes() 内の fill に factor(death) を指定しています。
geom_density() 内の alpha は不透過率を示しています。 scale_fill_manual() では自分で色を指定し(values)、それぞれの label も指定しています。
theme() 内では、凡例のタイトルやラベルの文字の体裁も変更できます。今回は 、legend.title と legend.text の引数を使って文字サイズを変更しています。
ggplot(nhefs01, aes(x = pack_years_n, fill = factor(death))) +
# 密度関数プロットの描画
geom_density(alpha = 0.6) +
theme_bw() +
# 塗りつぶし色を指定(ラベルも変更)
scale_fill_manual(values = c("#4169e1", "#ff6347"),
labels = c("Alive", "Dead")) +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 20),
legend.title = element_text(size = 18),
legend.text = element_text(size = 16)) +
labs(x = "Pack-Year", fill = "Death")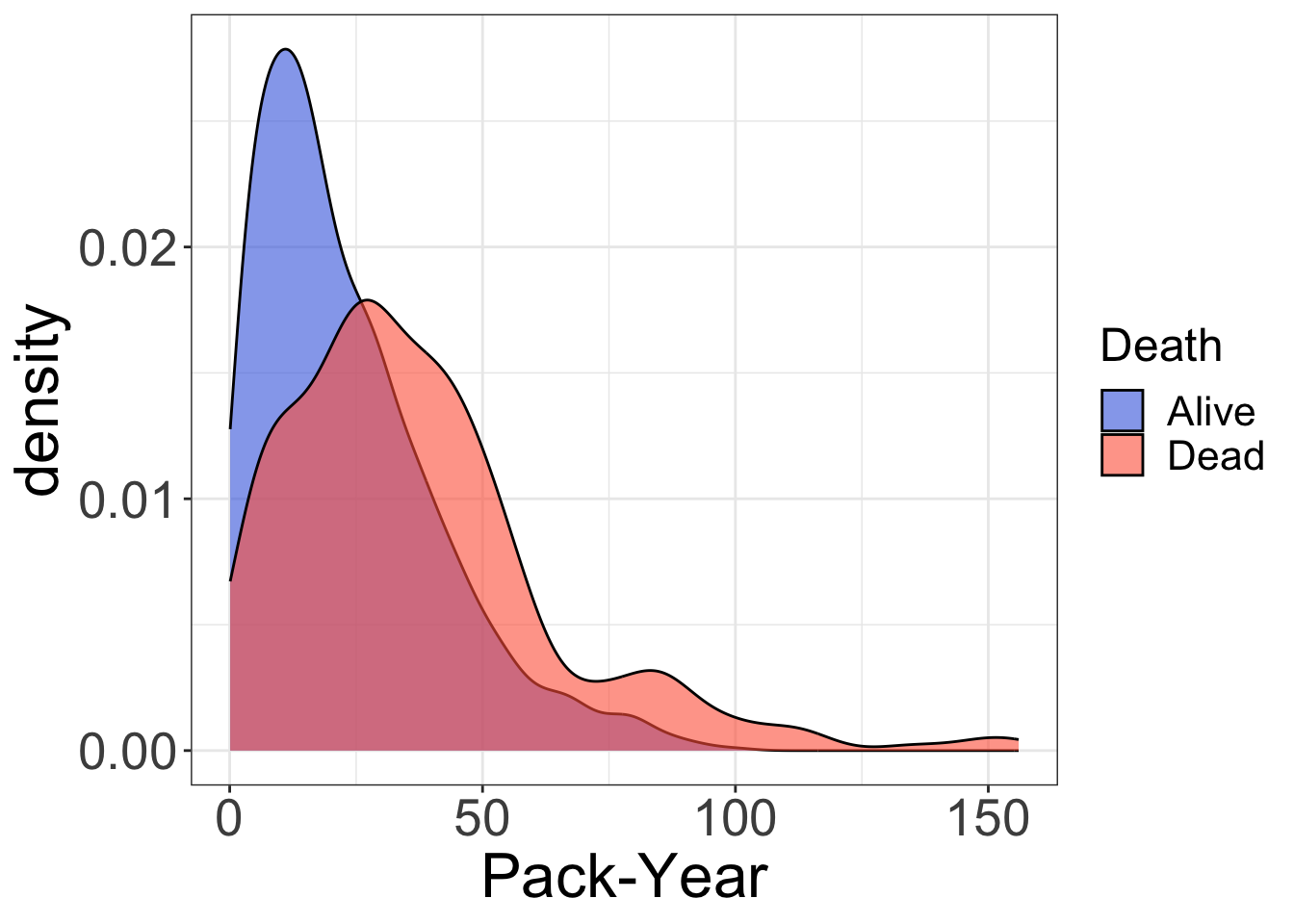
4.3 箱ひげ図
4.3.1 基本形
箱ひげ図には、geom_boxplot() を使用します。x軸に死亡の有無を、y軸にPack-Yearを配置して、死亡の有無で色の塗り分けをしていきます。
ggplot(nhefs01, aes(x = factor(death), y = pack_years_n, fill = factor(death))) +
# 箱ひげ図の描画
geom_boxplot()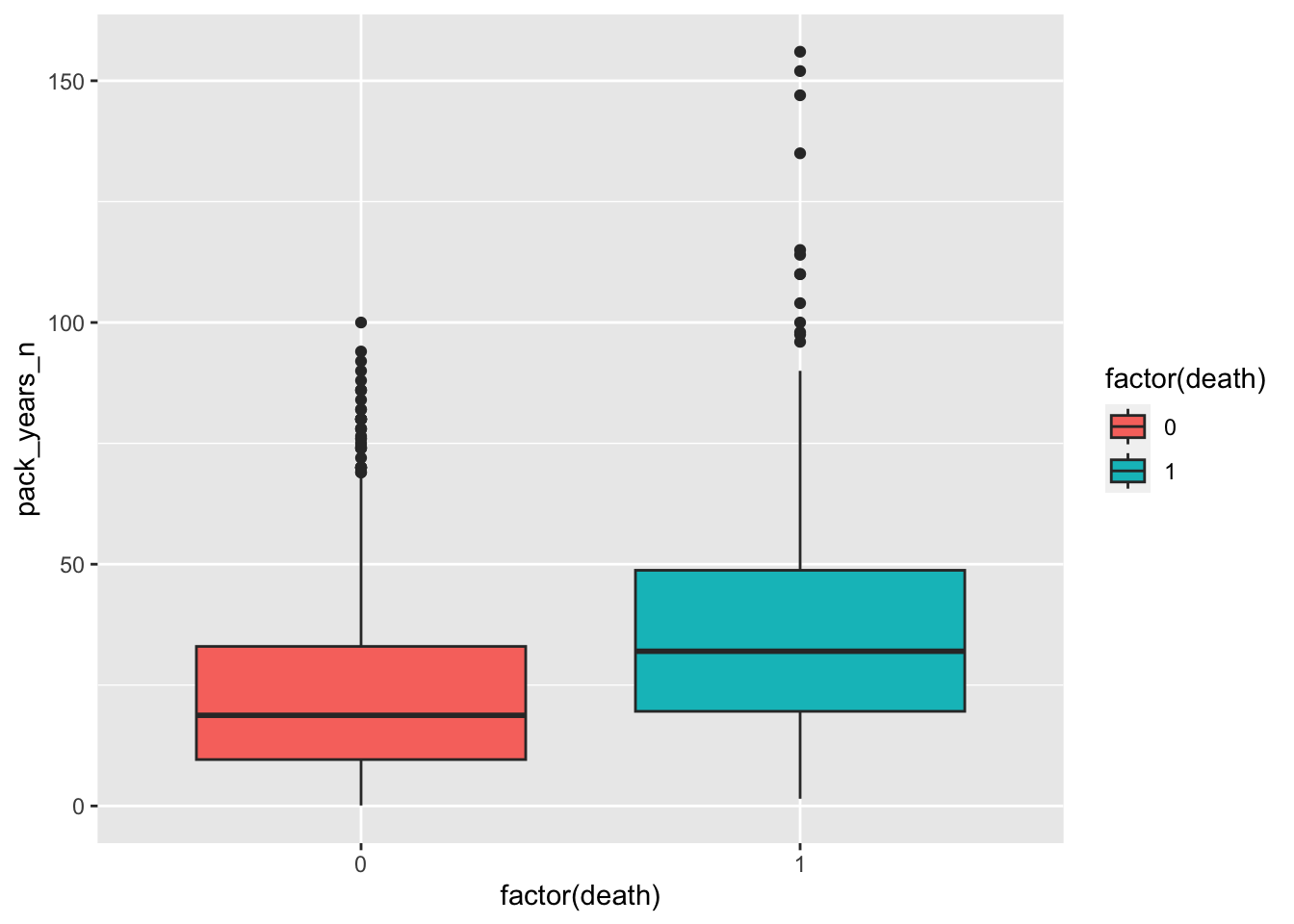
4.3.2 進化形1(箱の塗り分け)
scale_fill_manual() では自分で色を指定し(values)、それぞれの label も指定しています。
ggplot(nhefs01, aes(x = factor(death), y = pack_years_n, fill = factor(death))) +
geom_boxplot() +
# 箱の塗り分け
scale_fill_manual(values = c("#4169e1", "#ff6347"),
labels = c("Alive", "Dead"))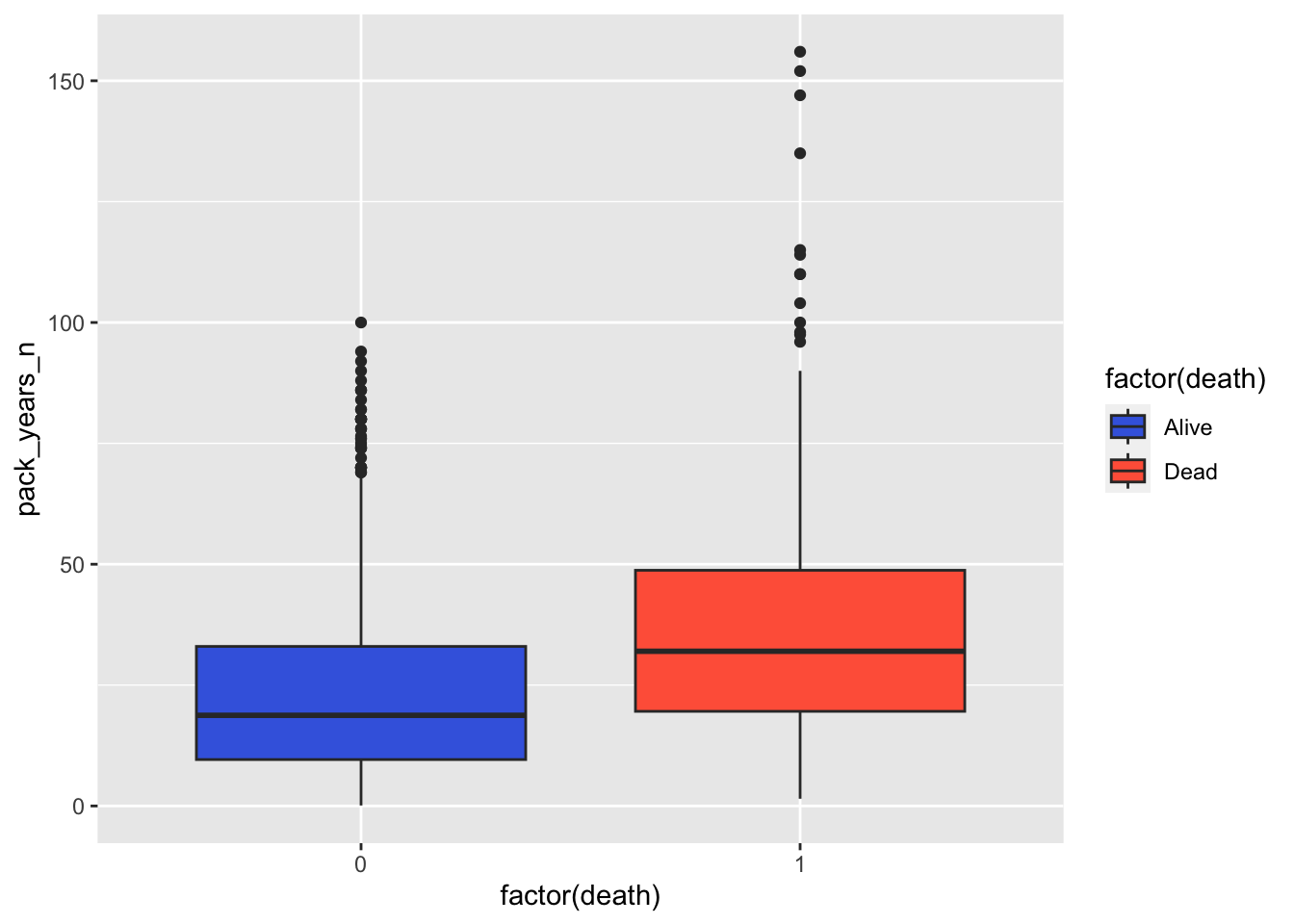
4.3.3 進化形2(背景と軸タイトル・テキストの体裁編集)
Part1でも使用した theme_bw() を使い、theme でフォントサイズを指定します。
ggplot(nhefs01, aes(x = factor(death), y = pack_years_n, fill = factor(death))) +
geom_boxplot() +
scale_fill_manual(values = c("#4169e1", "#ff6347"),
labels = c("Alive", "Dead")) +
# 背景の設定
theme_bw() +
# 軸タイトル・テキストの設定
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 20))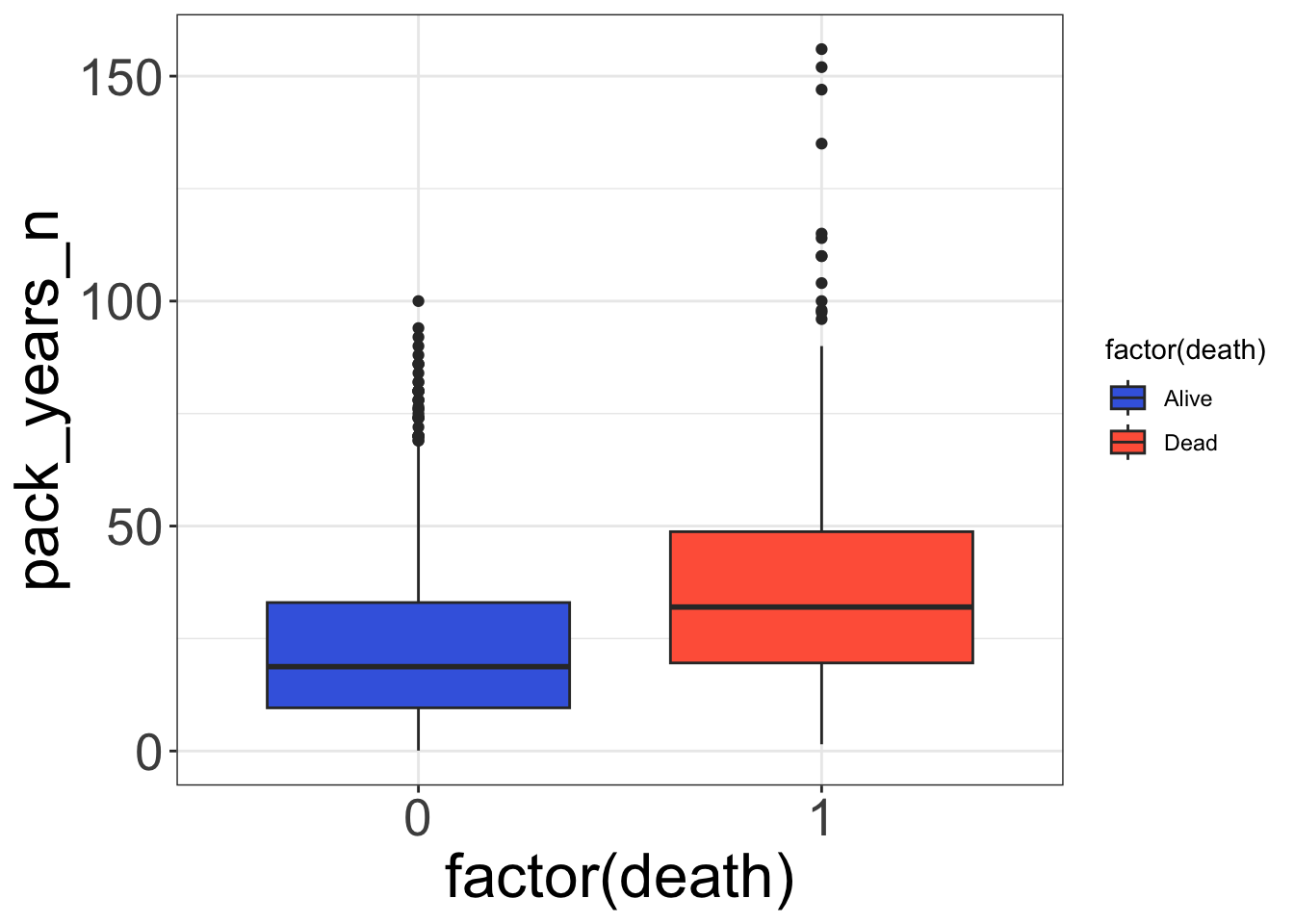
4.3.4 進化形3(凡例の削除と軸タイトルの変更)
凡例の設定は、theme() 内の引数 legend.position を使います。凡例を削除する場合には、"none" と指定します。
ggplot(nhefs01, aes(x = factor(death), y = pack_years_n, fill = factor(death))) +
geom_boxplot() +
scale_fill_manual(values = c("#4169e1", "#ff6347"),
labels = c("Alive", "Dead")) +
theme_bw() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 20),
legend.position = "none") +
# 軸のタイトルを変更
labs(x = "Death", y = "Pack-Year", fill = "Death")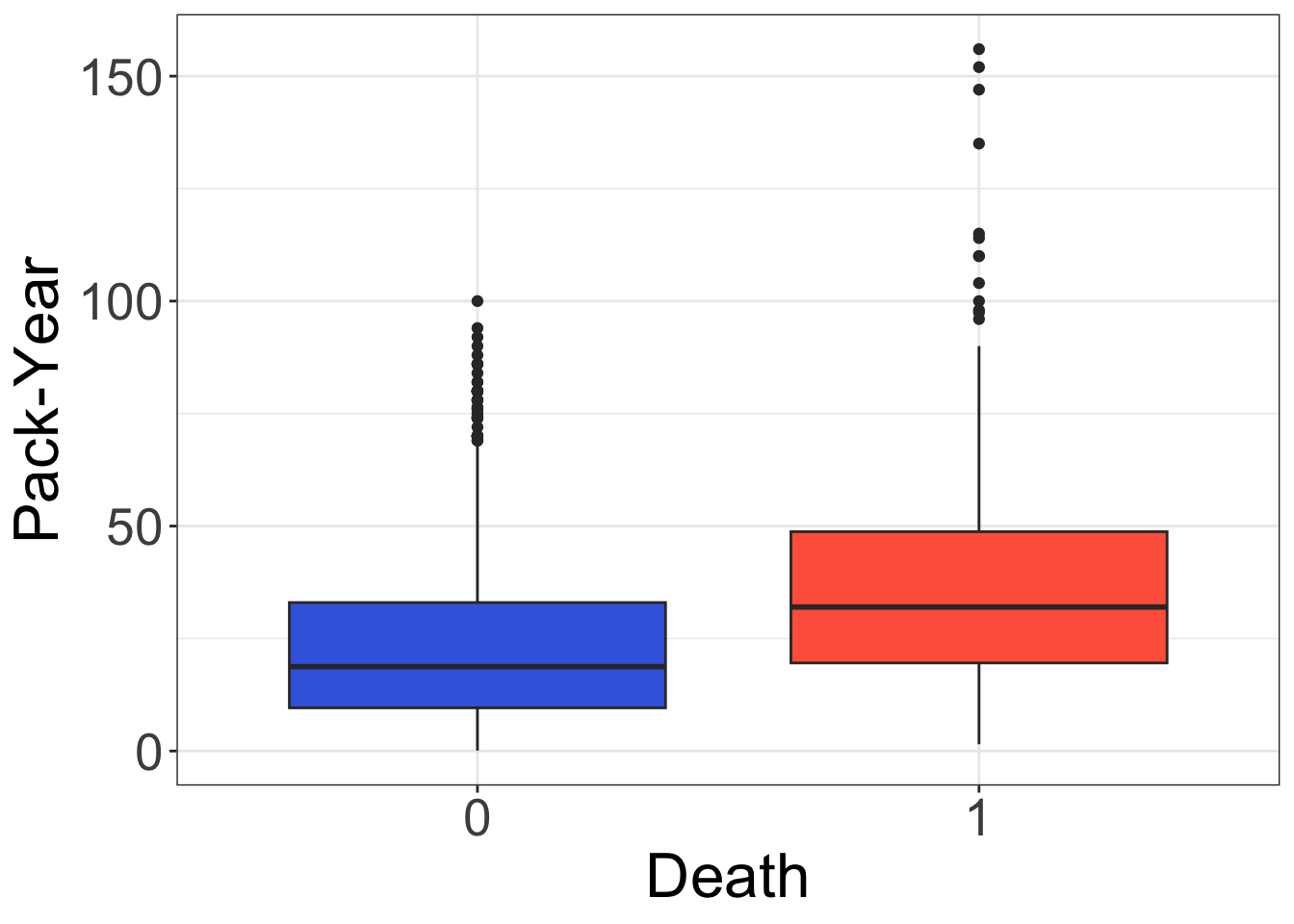
4.3.5 進化形4(軸ラベルの変更)
scale_x_discrete() では、x軸がカテゴリ変数の場合の軸の設定ができます。
ggplot(nhefs01, aes(x = factor(death), y = pack_years_n, fill = factor(death))) +
geom_boxplot() +
scale_fill_manual(values = c("#4169e1", "#ff6347"),
labels = c("Alive", "Dead")) +
theme_bw() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 20),
legend.position = "none") +
labs(x = "Death", y = "Pack-Year", fill = "Death") +
# x軸のラベルを変更
scale_x_discrete(labels = c("Alive", "Dead"))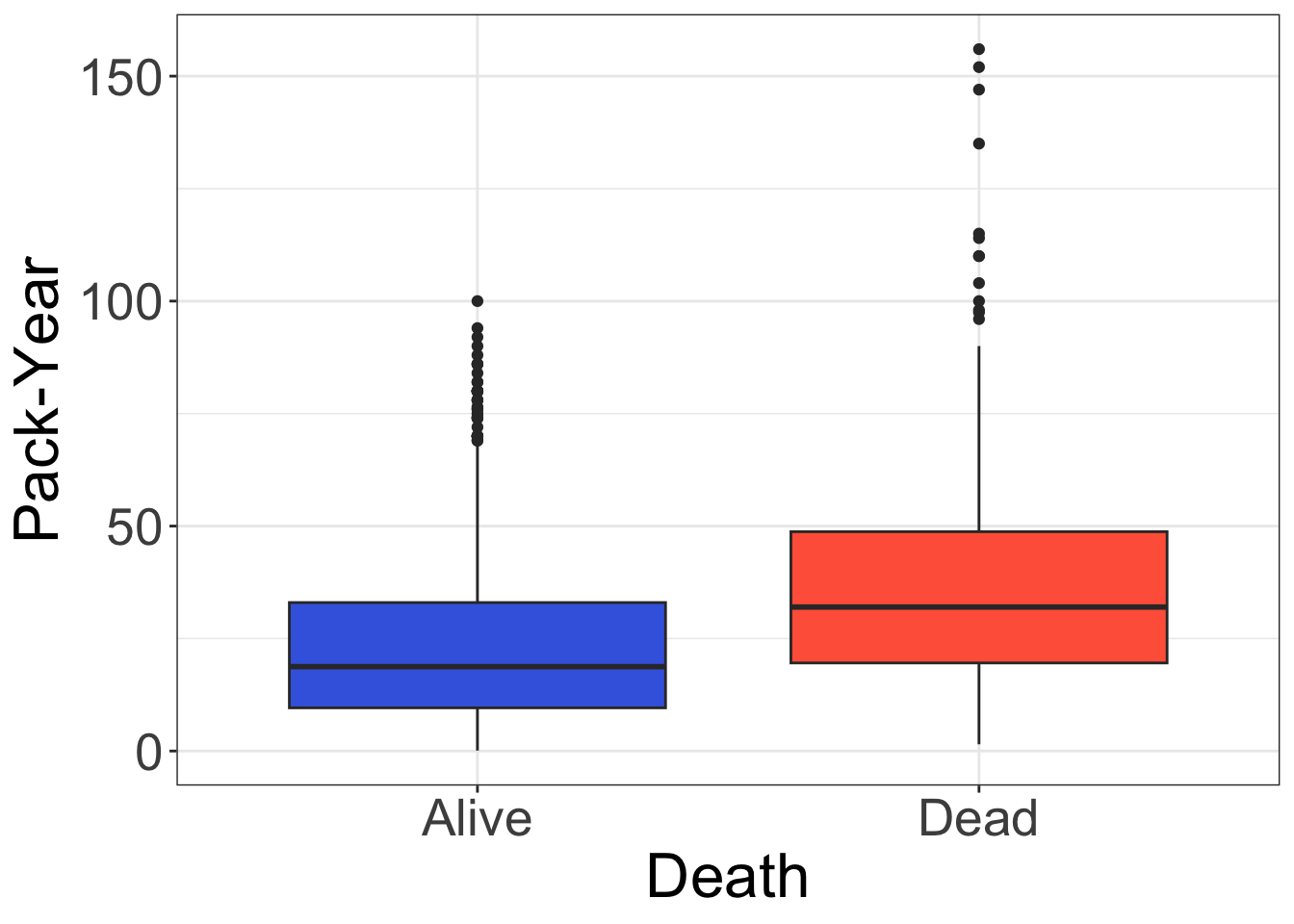
4.3.6 最終形(ジッタープロットの上乗せ)
ここに、geom.jitter() でドットプロットを上乗せします。geom_boxplot() 内の引数outlier.shape を NA にしておかないと、外れ値を二重にプロットしてしまうので、要注意です。
ggplot(nhefs01, aes(x = factor(death), y = pack_years_n, fill = factor(death))) +
geom_boxplot(outlier.shape = NA) +
# ジッタープロットの追加
geom_jitter(size = 2, alpha = 0.3, width = 0.3, color = "Black") +
scale_fill_manual(values = c("#4169e1", "#ff6347"),
labels = c("Alive", "Dead")) +
theme_bw() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 20),
legend.position = "none") +
labs(x = "Death", y = "Pack-Year", fill = "Death") +
scale_x_discrete(labels = c("Alive", "Dead"))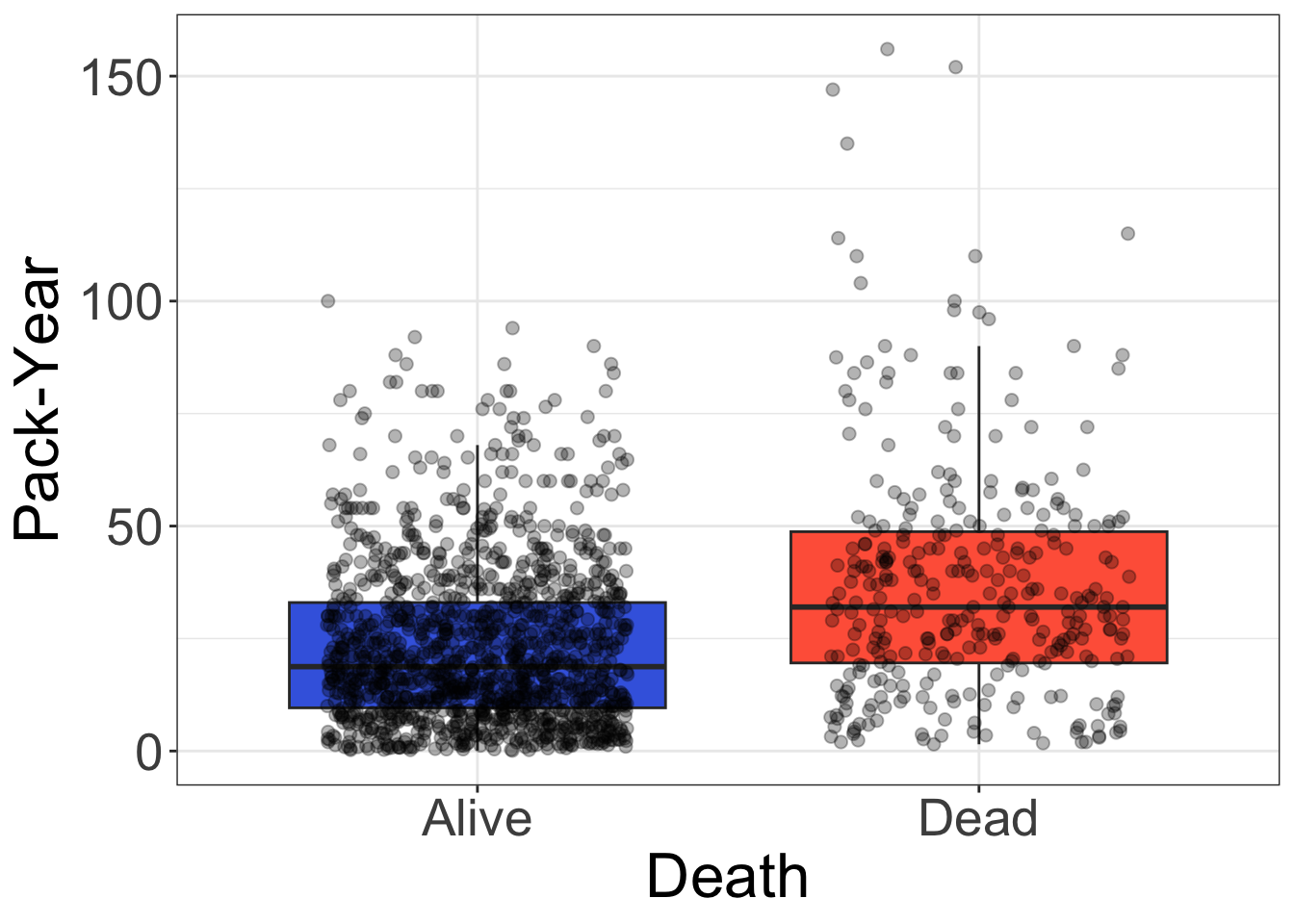
4.4 ドットプロット
連続変数について、特にサンプルサイズが小さい集団では、要約統計量だけでなく個別のデータをプロットするドットプロットが推奨されています4,5。
しかし、描画については注意する点がいくつかあるので、今回は美しいドットプロットを描くプロセスを追ってみたいと思います。
4.4.1 何も施していないドットプロット
基本的なドットプロットは、geom_point() で描くことができます6。箱ひげ図と同じ情報(x軸に死亡の有無を、y軸にPack-Year)を使用して描いてみましょう。
箱ひげ図と同じ変数で、
geom_point()を使用してドットプロットを描く点のサイズを変更する(
geom_point()内でsize = 6に設定します)
Code
ggplot(nhefs01, aes(x = factor(death), y = pack_years_n)) +
geom_point(size = 6)
4.4.2 色を塗り分けたドットプロット
次に、色の塗り分けをしていきます。
塗り分ける変数を指定する(
aes()内でfill = factor(death)を指定する)点の形を変更する(
geom_point()内でshape = 21に設定します)点の塗り分けを行う(
scale_fill_manual()を使用して色を指定します、箱ひげ図と同様です)
Code
# fill を追加
ggplot(nhefs01, aes(x = factor(death), y = pack_years_n, fill = factor(death))) +
# shape = 21を追加
geom_point(size = 6, shape = 21) +
# 点の塗りつぶしを追加
scale_fill_manual(values = c("#4169e1", "#ff6347"),
labels = c("Alive", "Dead"))
4.4.3 ポイントサイズを小さくしたドットプロット
先ほどのプロットでは、点と点の重なる部分が大きいので、点の大きさを小さくしましょう。
- 点のサイズを変更する(
geom_point()内でsize = 2に設定します)
Code
ggplot(nhefs01, aes(x = factor(death), y = pack_years_n, fill = factor(death))) +
# sizeを 2 に変更
geom_point(size = 2, shape = 21) +
scale_fill_manual(values = c("#4169e1", "#ff6347"),
labels = c("Alive", "Dead"))
4.4.4 透過率を大きくしたドットプロット
さらに、透過率をあげて、各点の視認性を向上していきます。
- 点の透過度を変更する(
geom_point()内でalpha = 0.6に設定します)
Code
ggplot(nhefs01, aes(x = factor(death), y = pack_years_n, fill = factor(death))) +
# 非透過度を60%に変更
geom_point(size = 2, shape = 21, alpha = 0.6) +
scale_fill_manual(values = c("#4169e1", "#ff6347"),
labels = c("Alive", "Dead"))
4.4.5 点を少しずらしたジッタープロット
今度は ジッターリングというテクニックを使用して、縦一列に並んでいた点を左右にずらしていきます。これにより、点同士の重なりが少なくなり、より各データの視認性を高くします。
ジッタープロットに変更する(
geom_point()の代わりに、geom_jitter()を使用します)ジッターリングの幅を指定する(
geom_jitter()内でwidth = 0.2と設定します)ジッタープロットの大きさや形、透過率は、
geom_point()のものをそのまま引き継いでください
Code
ggplot(nhefs01, aes(x = factor(death), y = pack_years_n, fill = factor(death))) +
# widthで配置をずらす
geom_jitter(size = 2, shape = 21, alpha = 0.6, width = 0.2) +
scale_fill_manual(values = c("#4169e1", "#ff6347"),
labels = c("Alive", "Dead"))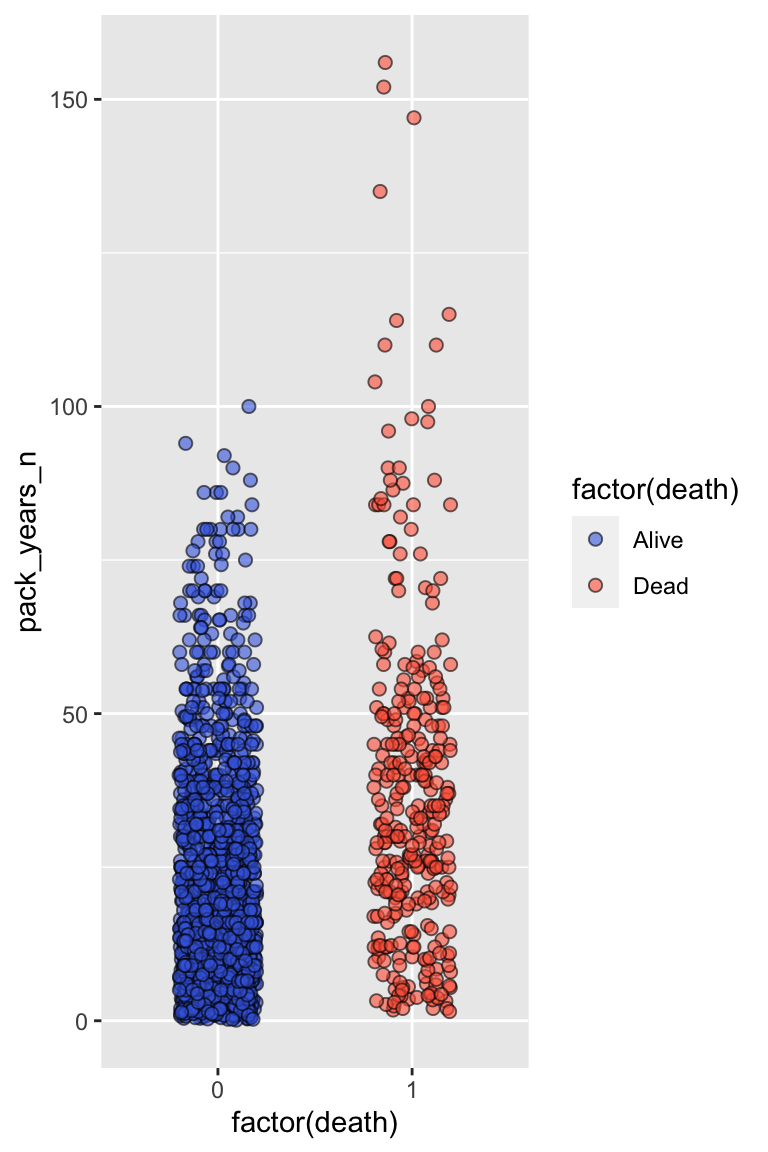
4.4.6 確率分布にもとづいて点をずらしたビースウォームプロット
不規則に点をずらしていたグラフに比べてジッタープロットから、確率分布にしたがって点をずらすビースウォームプロットに変更します。これにより、もう少し分布が把握しやすくなります。
geom_beeswarm() では、width 引数の代わりに、cex 引数で点の広がりを指定できますが、今回はデフォルトのままで構いません。
- ビースウォームプロットに変更する(
geom_jitter()の代わりに、geom_beeswarm()を使用します)
Code
ggplot(nhefs01, aes(x = factor(death), y = pack_years_n, fill = factor(death))) +
# ビースウォームプロットの描画
geom_beeswarm(size = 2, alpha = 0.6, shape = 21) +
scale_fill_manual(values = c("#4169e1", "#ff6347"),
labels = c("Alive", "Dead"))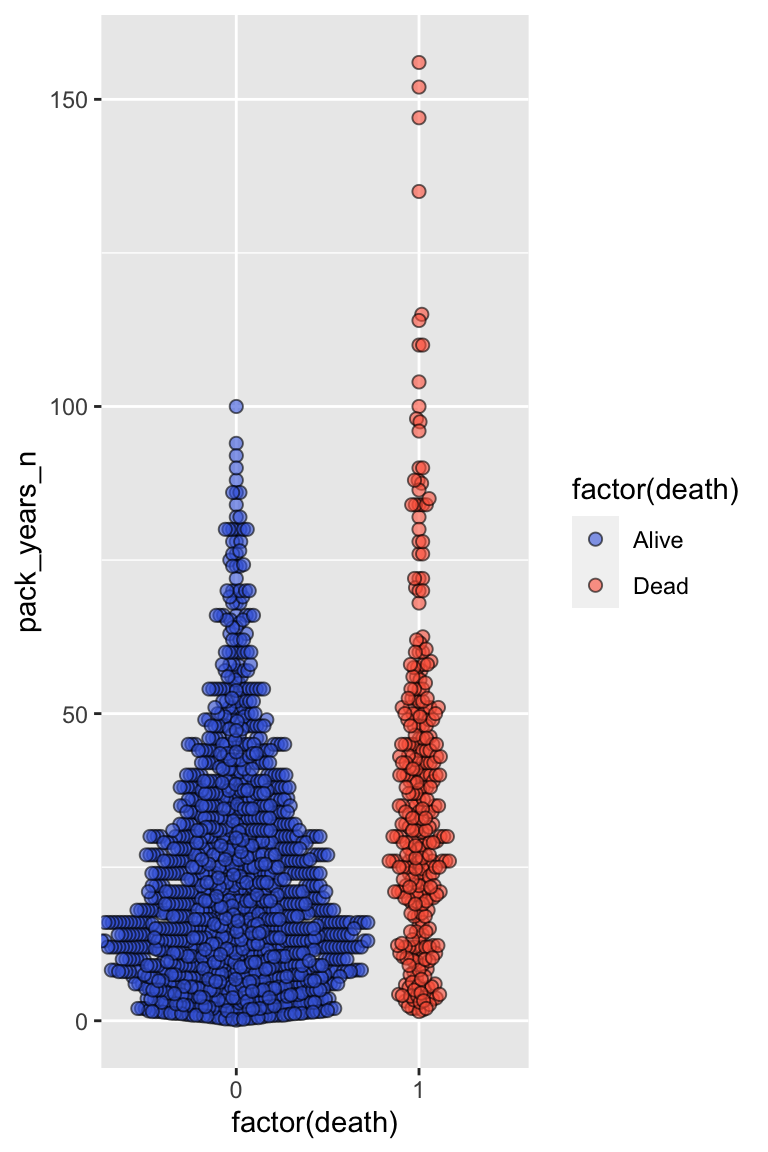
4.4.7 最終形(背景や軸の体裁編集)
箱ひげ図でも行ったように、背景の変更、x, y軸のタイトル編集、x軸のラベル編集、凡例の削除を行います。
- あえてヒントなしで(箱ひげ図のものを参考にしてやってみてください)
Code
ggplot(nhefs01, aes(x = factor(death), y = pack_years_n, fill = factor(death))) +
# ビースウォームプロットの描画
geom_beeswarm(size = 2, alpha = 0.6, shape = 21) +
scale_fill_manual(values = c("#4169e1", "#ff6347"),
labels = c("Alive", "Dead")) +
theme_bw() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 20),
legend.position = "none") +
scale_x_discrete(labels = c("Alive", "Dead")) +
labs(x = "Mortality", y = "Pack-Year")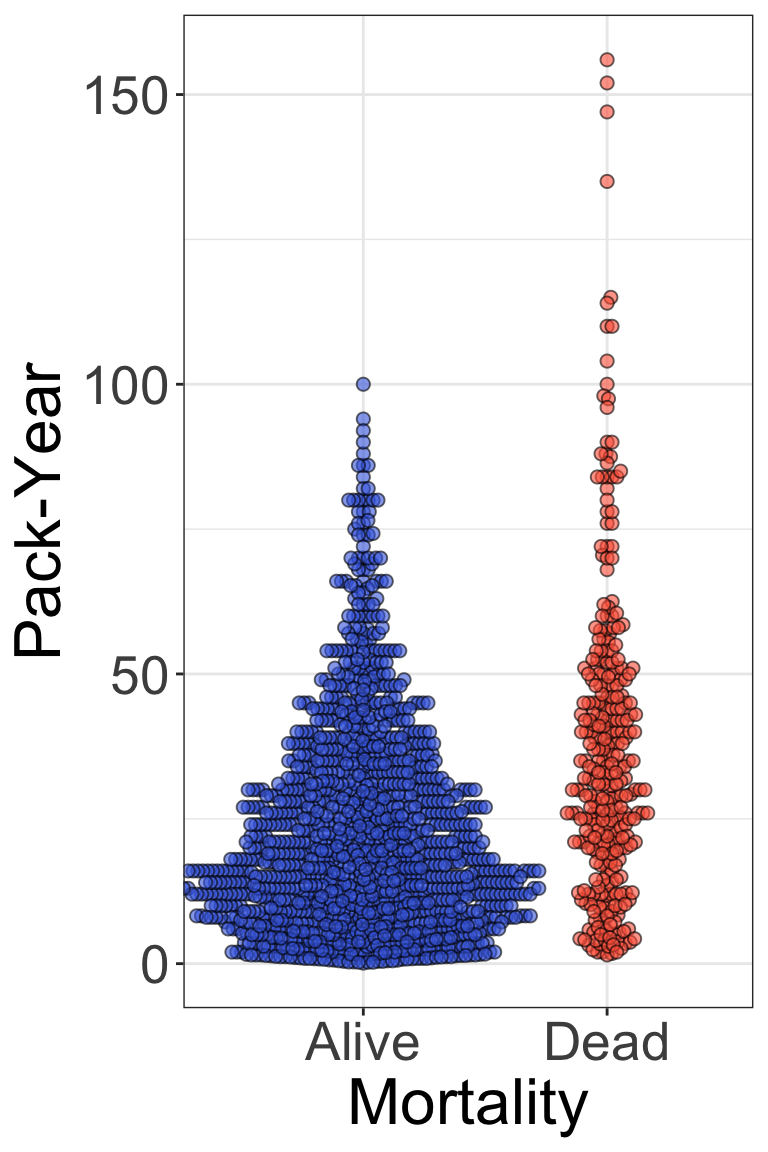
4.5 レインクラウドプロット
これまでみてきた連続変数の可視化について、分布・要約統計量・各データの全てを網羅的に可視化する方法がレインクラウドプロットです。
現時点では、raincloudplotsパッケージが最も使いやすいと思いますが、データの整理など少し不便な点もあります。
# remotes::install_github('jorvlan/raincloudplots')
# 生存のみのデータセットを作成
nhefs_alive <- nhefs01 |>
filter(death == 0)
# 死亡のみのデータセットを作成
nhefs_dead <- nhefs01 |>
filter(death == 1)
# 上で作成したデータのpack_year_nを入力として使用する
df_1x1 <- data_1x1(
array_1 = nhefs_alive$pack_years_n,
array_2 = nhefs_dead$pack_years_n)
raincloud_1x1(
data = df_1x1,
colors = (c("#696969", "#696969")),
fills = (c("#4169e1", "#ff6347")),
size = 1.5,
alpha = .8,
ort = 'h') +
scale_x_continuous(breaks = c(1.2, 2.2),
labels = c("Alive", "Dead"),
limits = c(0.7, 3)) +
labs(x = "Mortality", y = "Pack-Year") +
theme_bw() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 20))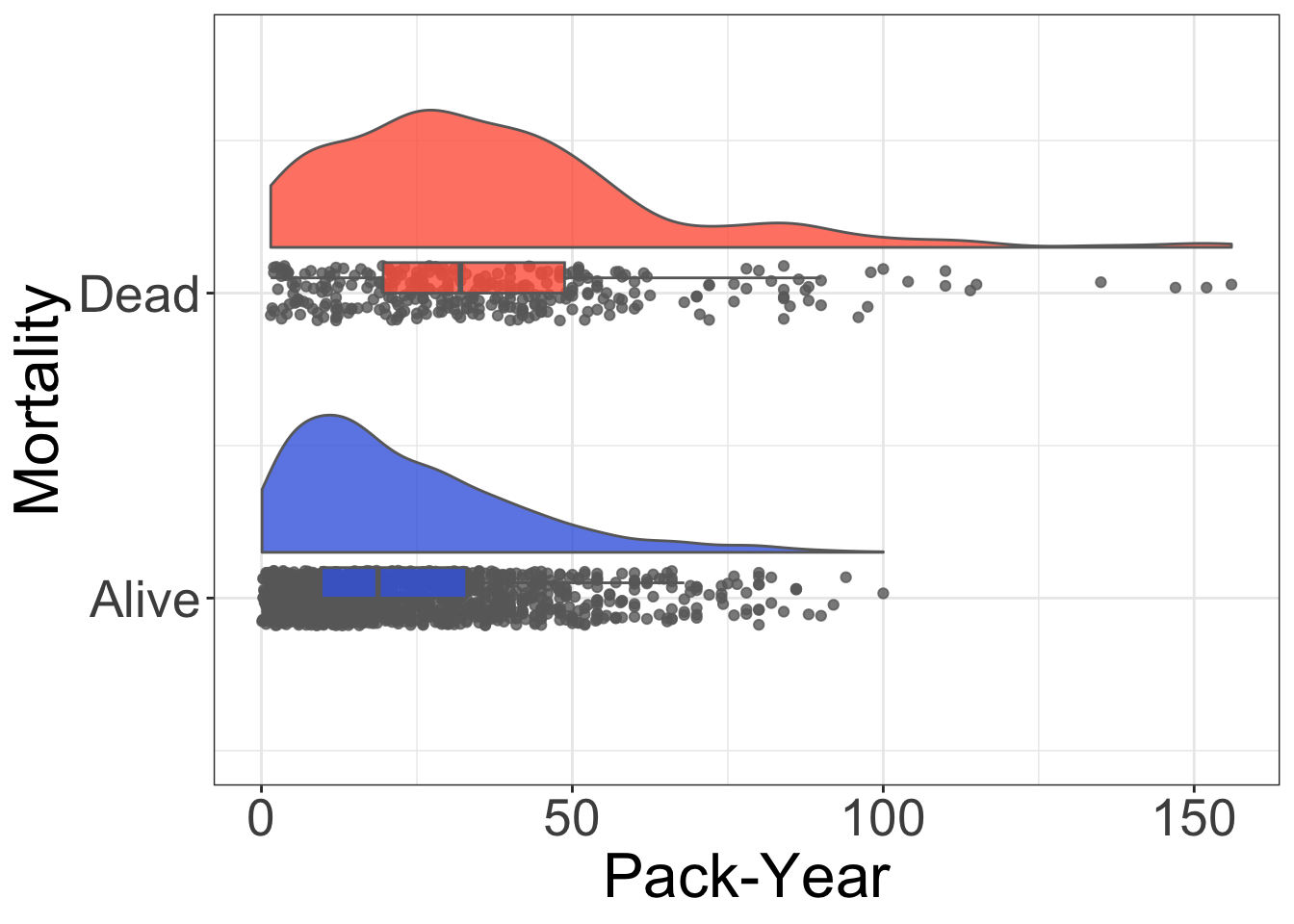
5 Nominal variable(名義変数)
ここでは、複数のカテゴリを持つ marital (婚姻状況)や education (教育年数)を用いて、名義変数(一変量および二変量)の可視化方法を確認していきます。
5.1 棒グラフ
まずは、基本的な棒グラフです。一変量の数え上げには、棒グラフを使用することをお勧めします(円グラフよりも大小の見分けがしやすいです)。
5.1.1 ラベルなし
geom_bar() を使用して描きますが、stat 引数に count と指定することがポイントです7。
ggplot(nhefs01, aes(x = factor(marital))) +
# 棒グラフの描画
geom_bar(stat = "count") +
theme_bw() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 20)) +
labs(x = "Marital status")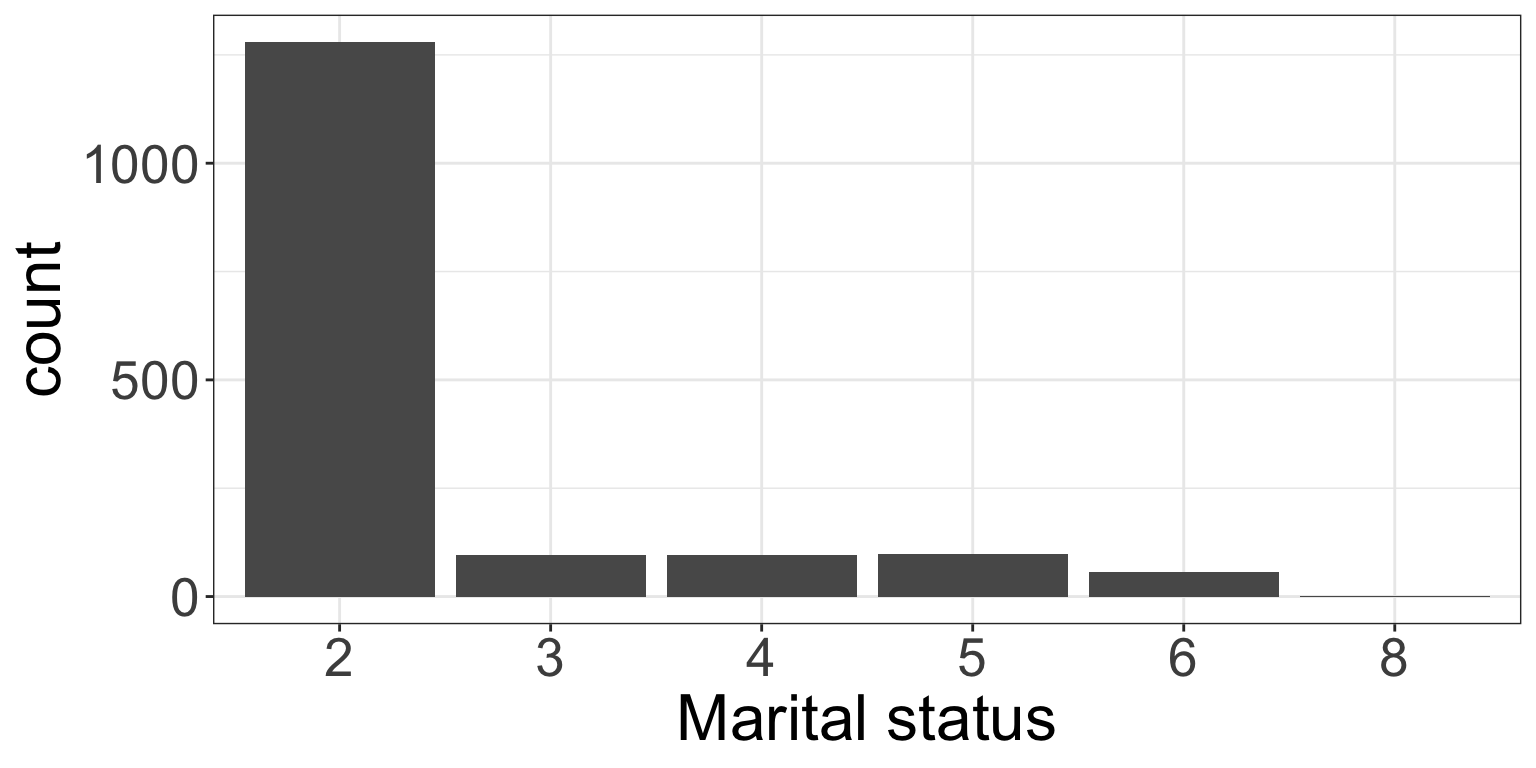
「可視化は面倒だ」という人は、table() を使用して少なくともカテゴリごとの人数は確認しておきましょう。
table(nhefs01$marital)
2 3 4 5 6 8
1279 97 96 99 57 1 5.1.2 ラベルあり
先ほどの棒グラフは、チーム内にデータを共有する場合や欠測の確認をする場合には問題ありませんが、外部に出すものとしては、ラベルがなく不十分(不親切)でした。
そこで変数情報を一部改訂して、再度可視化に臨みます。
nhefs02 <- nhefs01 |>
#変数情報(ラベル)の変更
mutate(marital = factor(marital,
levels = c(1,2,3,4,5,6,8),
labels = c("Under17",
"Married",
"Widowed",
"Never",
"Divorced",
"Separated",
"Unknown")))
ggplot(nhefs02, aes(x = marital)) +
geom_bar(stat = "count") +
theme_bw() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 18)) +
labs(x = "Marital status")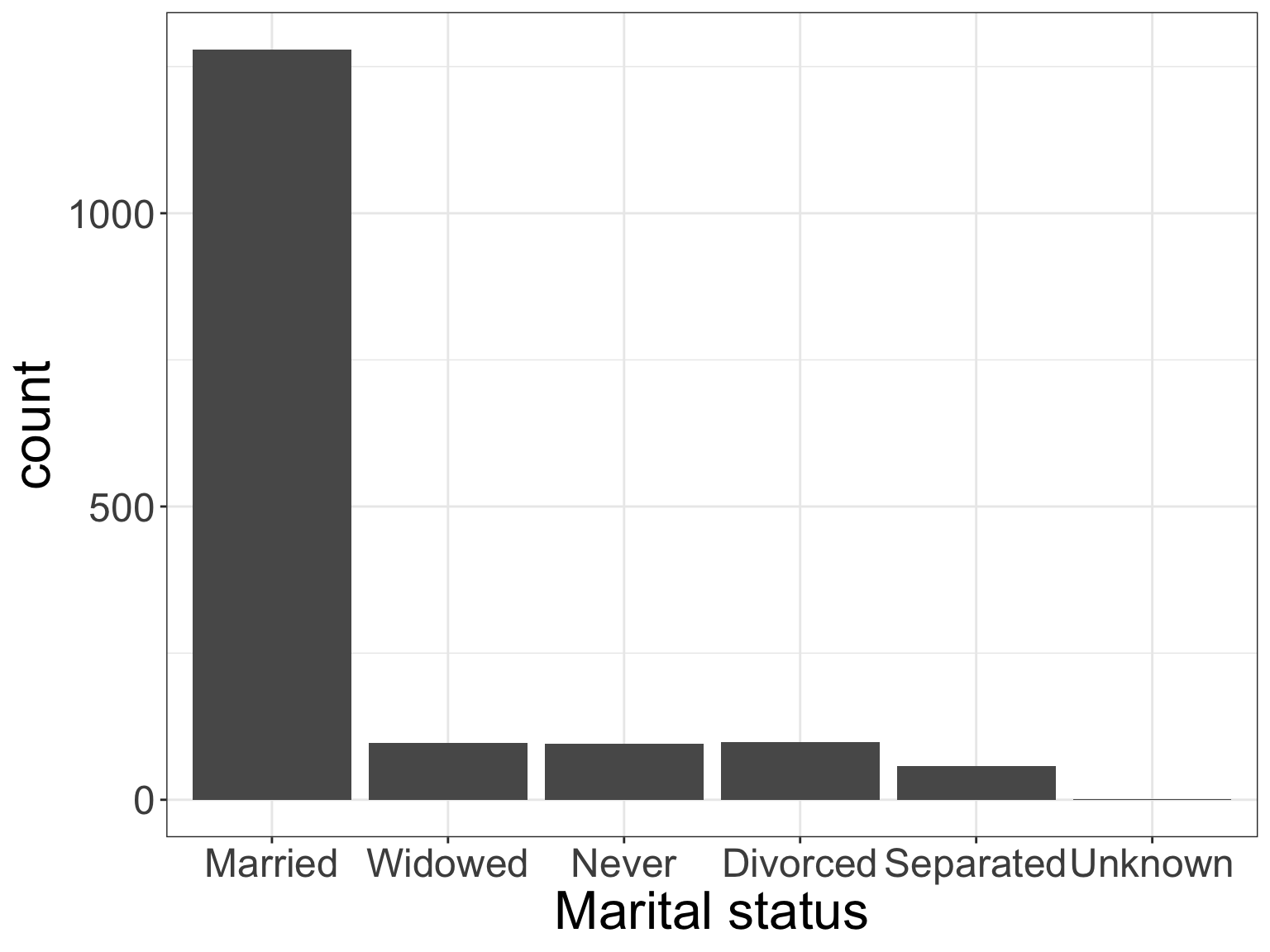
5.2 横並び棒グラフ
先ほどは、一変量の棒グラフでした。今度は Part 3 で使用する death のアウトカムで層別化した教育歴 education を確認します。
まずは、データセットの編集をします。
nhefs03 <- nhefs01 |>
mutate(education = factor(education,
labels = c("8th grade or less",
"HS dropout",
"HS",
"College dropout",
"College or more")),
death = factor(death,
labels = c("Alive", "Dead")))5.2.1 一変量の棒グラフ
先ほど、婚姻状況の可視化で行ったものを参考にして、同じように教育歴 education について一変量の棒グラフをまず作りましょう。
5.1.1 を参考にやってみましょう
x軸のタイトルは、Educationとします
Code
ggplot(nhefs03, aes(x = education)) +
geom_bar(stat = "count") +
theme_bw() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 18)) +
labs(x = "Education")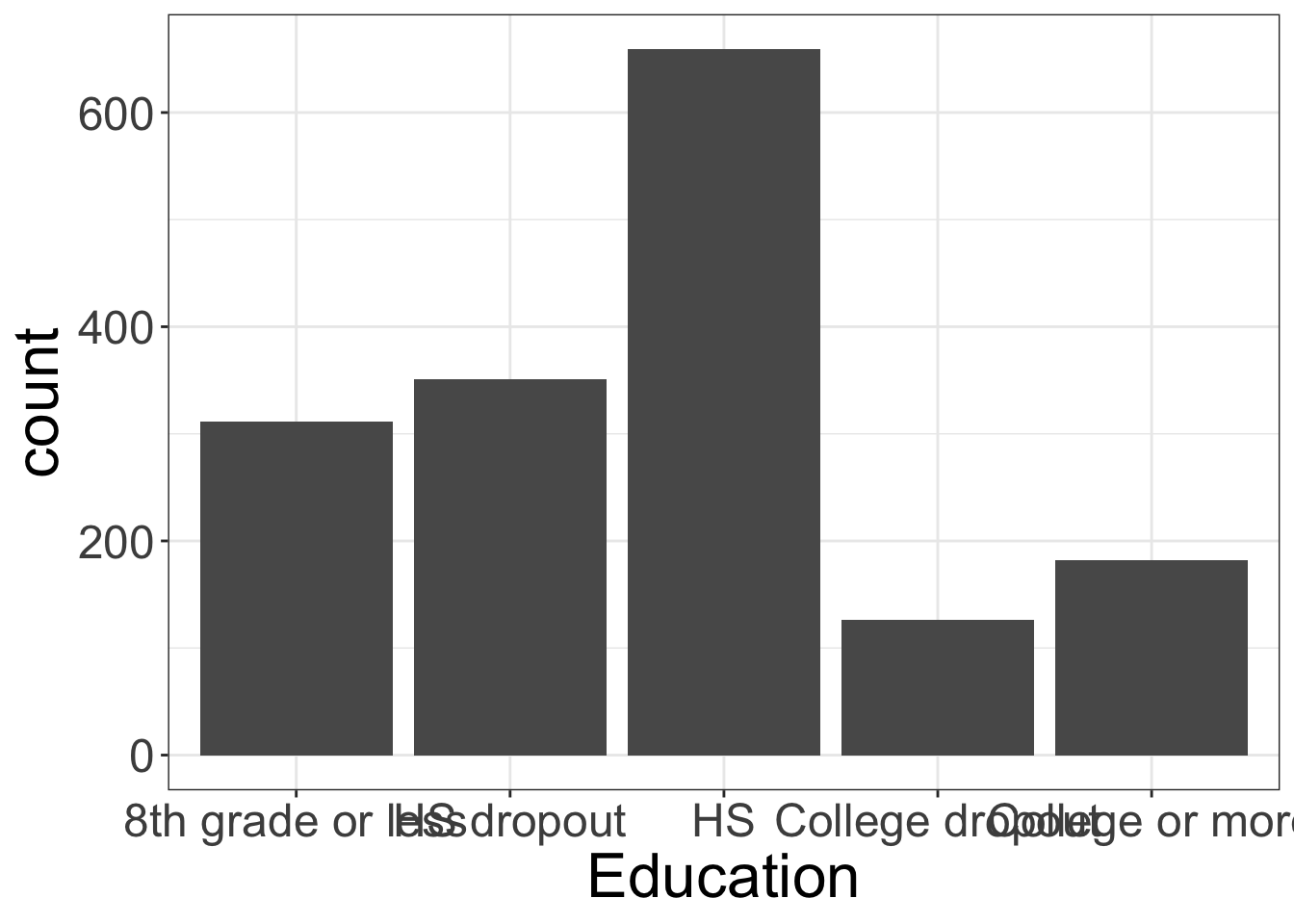
5.2.2 横並びの棒グラフ1
一変量の棒グラフからの横並びの棒グラフへと変身させていきます。
層別化する変数を指定します(
aes()内でfill = deathを指定します)棒グラフから横並び棒グラフへ変身させます(
geom_bar()内でposition = "dodge"を指定します)凡例のタイトルは、Deathとします(
labs()内でfill = "Death"を指定します)
Code
ggplot(nhefs03, aes(x = education, fill = death)) +
# 横並び棒グラフの描画
geom_bar(stat = "count", position = "dodge") +
theme_bw() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 18)) +
labs(x = "Education", fill = "Death")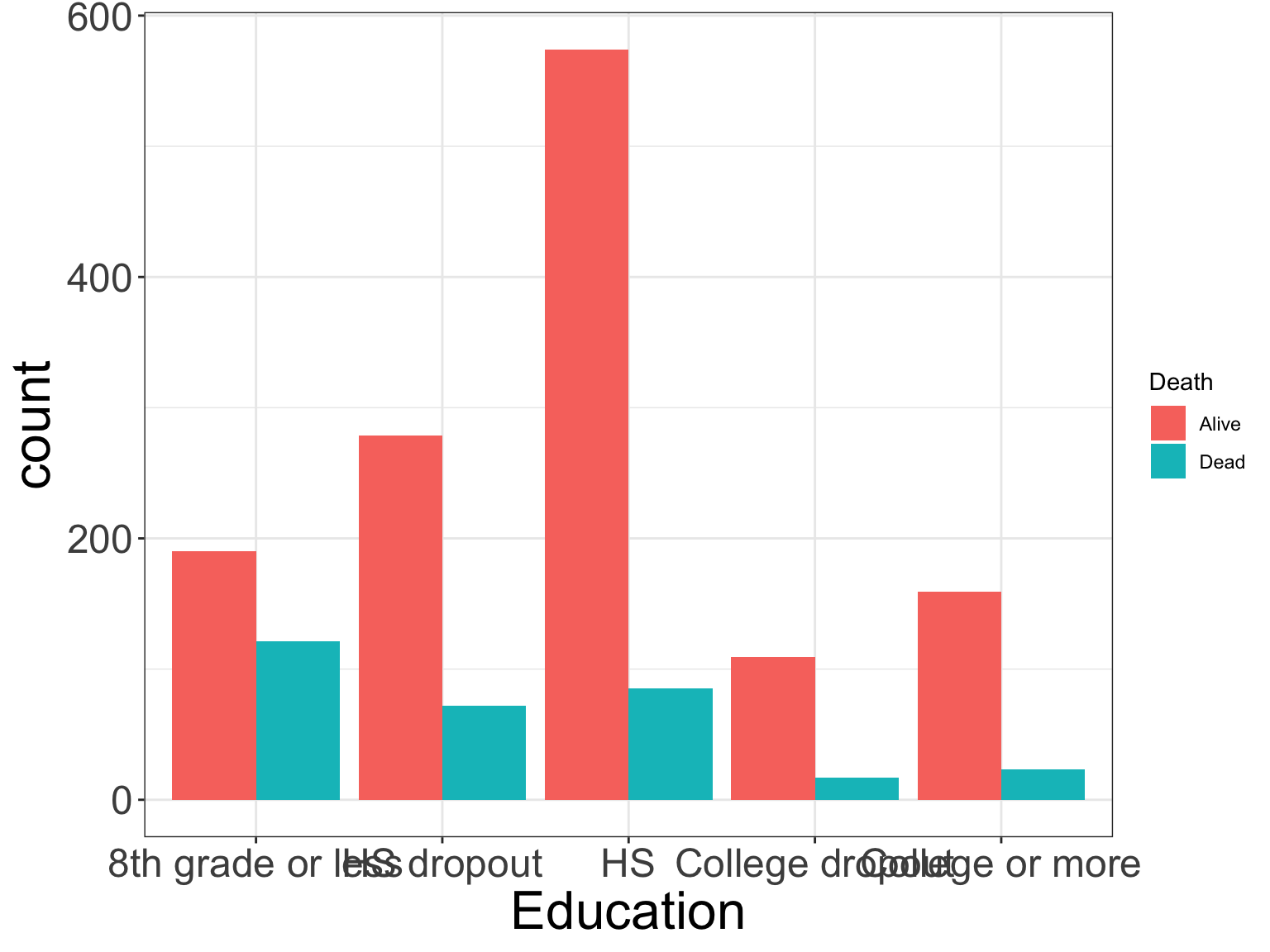
5.2.3 横並びの棒グラフ2(塗り分けの指定)
棒グラフの色を変更したいと思います。ここでは、これまでの方法と異なり、Rの既存のパレットを使ってみましょう。
カラーパレットについては、RColorBrewer のものが一般的によく使用されますが、描画する変数のタイプ(数値型、因子型など)に応じて適切なパレットを使用する必要があります。
COLORBREWER 2.0 を確認してみる
| 変数の種類 | 適したカラースケール |
|---|---|
| 定性データ | RColorBrewer (Set2) など |
| 連続データ(一方向) | RColorBrewer (Blues) など |
| 連続データ(両方向) | RColorBrewer (RdBu) など |
- 点の塗り分けを行う(
scale_fill_brewer()内に、palette = "Dark2"と指定します)
Code
ggplot(nhefs03, aes(x = education, fill = death)) +
geom_bar(stat = "count", position = "dodge") +
# パレットの指定
scale_fill_brewer(palette = "Dark2") +
theme_bw() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 18)) +
labs(x = "Education", fill = "Death")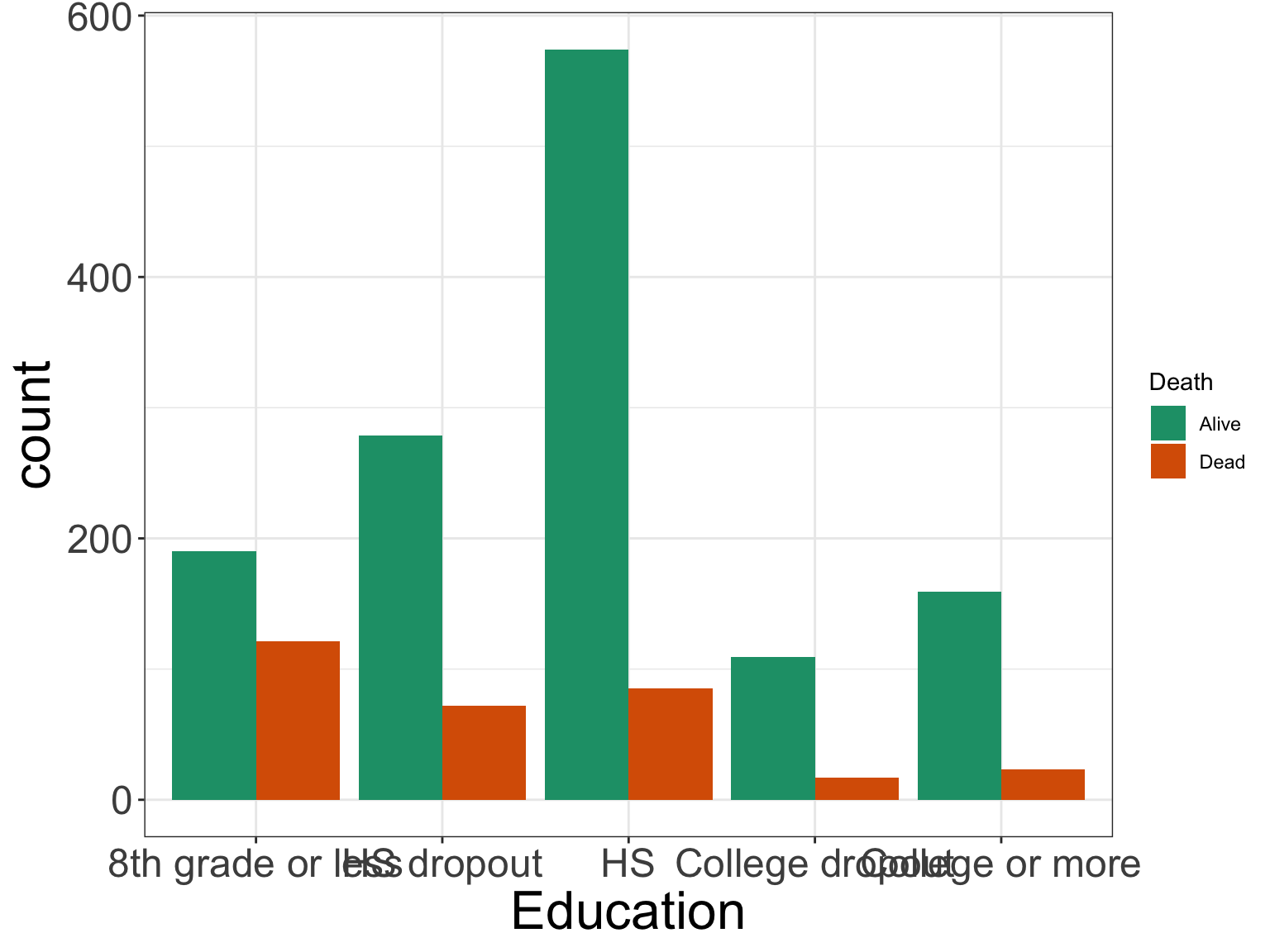
5.2.4 横並びの棒グラフ3(凡例の位置の変更)
ここから細かい体裁の編集に入ります。まずは、凡例の位置を上部に持っていきます。これにより、左側に Alive 、右側に Dead が並び、凡例と棒グラフの並びが対応します。
- 凡例の位置を変更する(
theme()内でlegend.position = "top"と指定します)
Code
ggplot(nhefs03, aes(x = education, fill = death)) +
geom_bar(stat = "count", position = "dodge") +
scale_fill_brewer(palette = "Dark2") +
theme_bw() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 18),
# 凡例の編集(上部に表示するなど)
legend.position = "top",
legend.title = element_text(size = 18),
legend.text = element_text(size = 16)) +
labs(x = "Education", fill = "Death")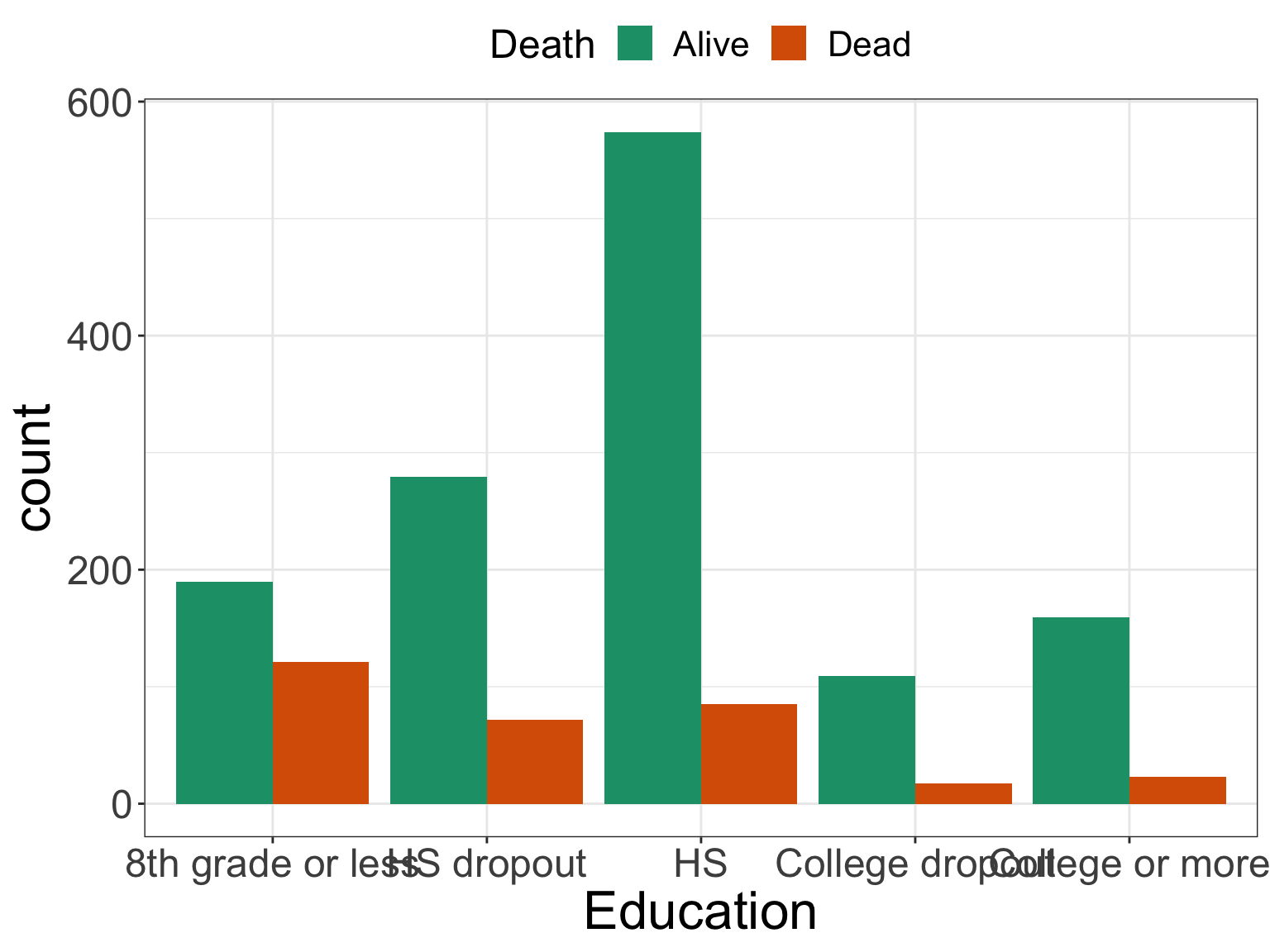
5.2.5 横並びの棒グラフ4(x軸のラベルの回転)
よく見ると、x軸のカテゴリ名が重なっていて、分かりづらくなっています。これを解消するために、x軸の変数名を回転します。
- x軸の文字を回転する(
theme()内でaxis.text.x = element_text(angle = 45, hjust = 1)を指定します)
Code
ggplot(nhefs03, aes(x = education, fill = death)) +
geom_bar(stat = "count", position = "dodge") +
scale_fill_brewer(palette = "Dark2") +
theme_bw() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 18),
# x軸の文字を45度に傾ける
axis.text.x = element_text(angle = 45, hjust = 1),
legend.position = "top",
legend.title = element_text(size = 18),
legend.text = element_text(size = 16)) +
labs(x = "Education", fill = "Death")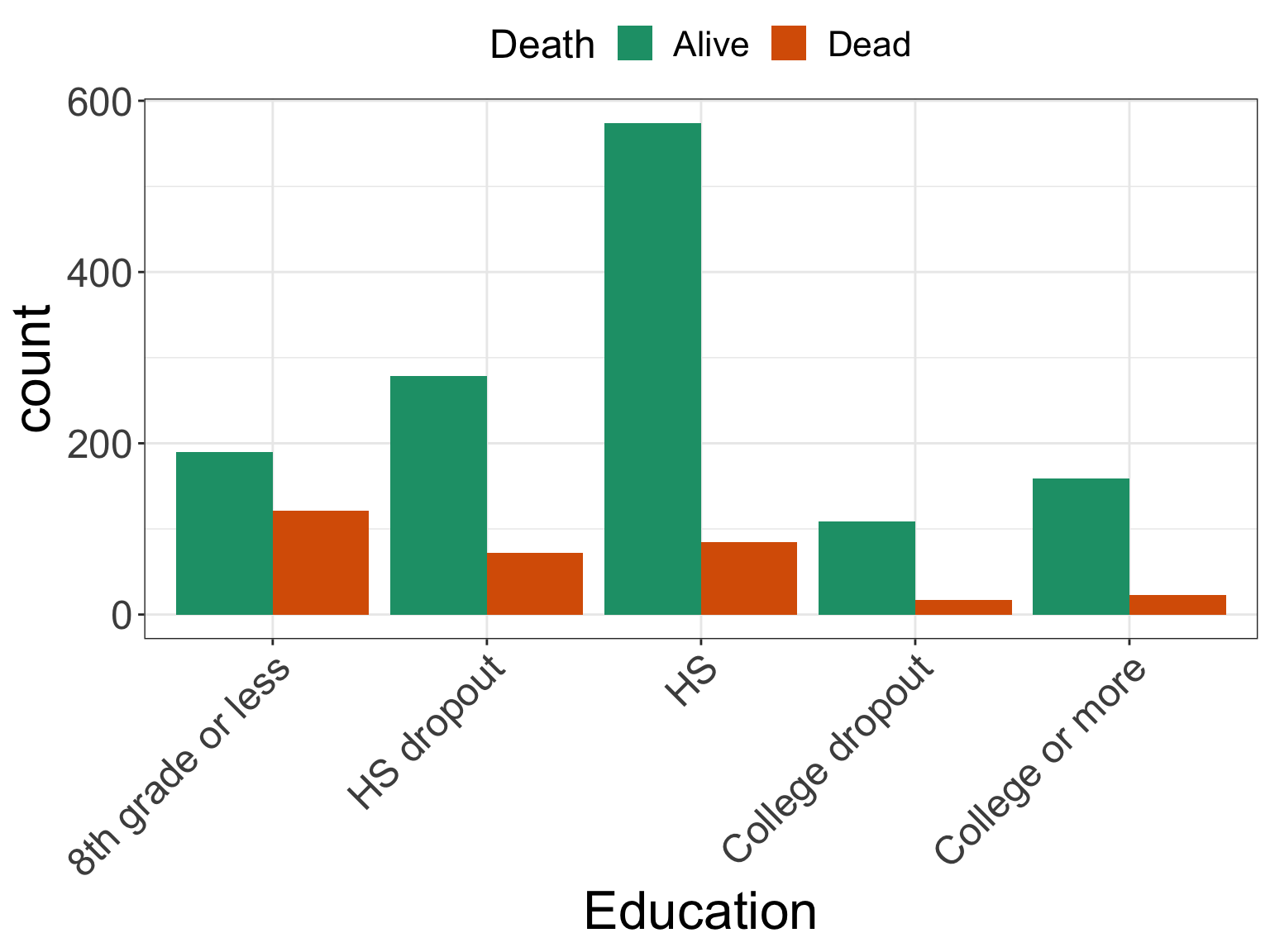
5.3 100%積み上げ棒グラフ
今度は絶対数ではなく、割合を確認してみたいので、100%積み上げ棒グラフを使用します。geom_bar() 内の position 引数に fill を指定することで、描くことができます。
事前準備として、factor() の levels を使って変数の順番を入れ替えています。
また、今回は coord_flip() で、x軸とy軸をひっくり返しています。
nhefs_100_bar <- nhefs03 |>
mutate(death = factor(death,
levels = c("Dead", "Alive")))
ggplot(nhefs_100_bar, aes(x = education, fill = death)) +
# 100%積み上げ棒グラフの描画
geom_bar(stat = "count", position = "fill") +
scale_fill_manual(values = c("#ff6347", "#4169e1")) +
# y軸を%表記にする
scale_y_continuous(labels = scales::percent_format()) +
# x軸とy軸を入れ替える
coord_flip() +
theme_bw() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 20),
legend.position = "top",
legend.title = element_text(size = 18),
legend.text = element_text(size = 16)) +
labs(x = "Education", y = "Percentage", fill = "Death") +
# 凡例も入れ替える
guides(fill = guide_legend(reverse = TRUE))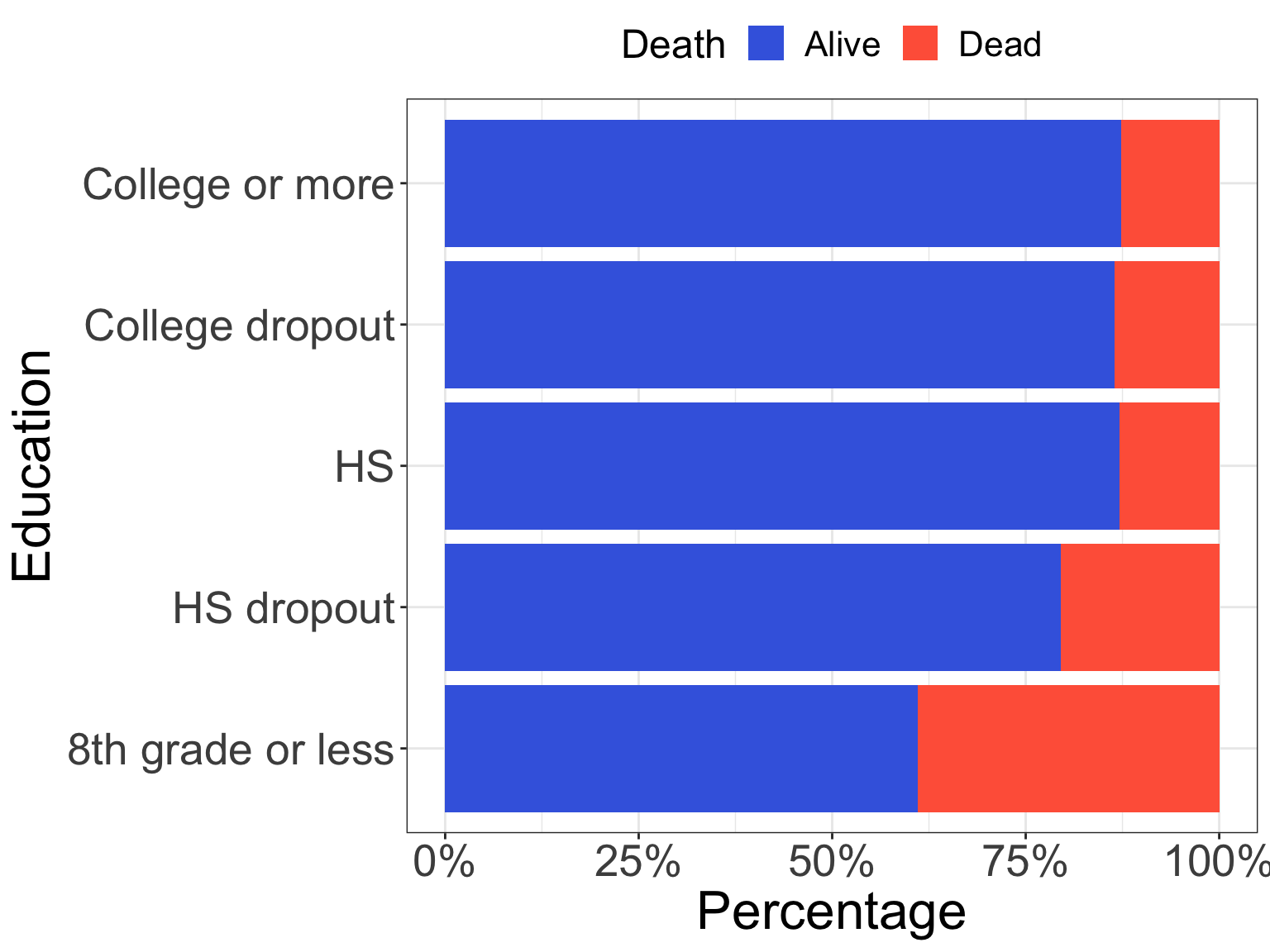
6 Multivariable(多変量)
Part 3 で利用する変数どうしの関係を可視化したいと思います。曝露変数(Pack-Year)と結果変数(Death)の列に注目して確認するようにしましょう。
今回は、GGally パッケージの ggpairs() を使用しています。
nhefs04 <- nhefs03 |>
# カテゴリー化、ラベリング
mutate(
death = factor(death,
labels = c("Alive", "Dead")),
sex = factor(sex,
labels = c("Male", "Female")),
race = factor(race,
labels = c("White", "Black or other")),
education = factor(education,
labels = c("8th grade or less",
"HS dropout",
"HS",
"College dropout",
"College or more")),
exercise = factor(exercise,
labels = c("Much exercise",
"Moderate exercise",
"LIttle or no excercise")))
ggpairs(nhefs04,
columns = c("death", "sex", "age", "race", "education", "exercise", "pack_years_n"),
columnLabels = c("Death", "Sex", "Age", "Race", "Education", "Exercise", "Pack-Year")) +
theme_bw()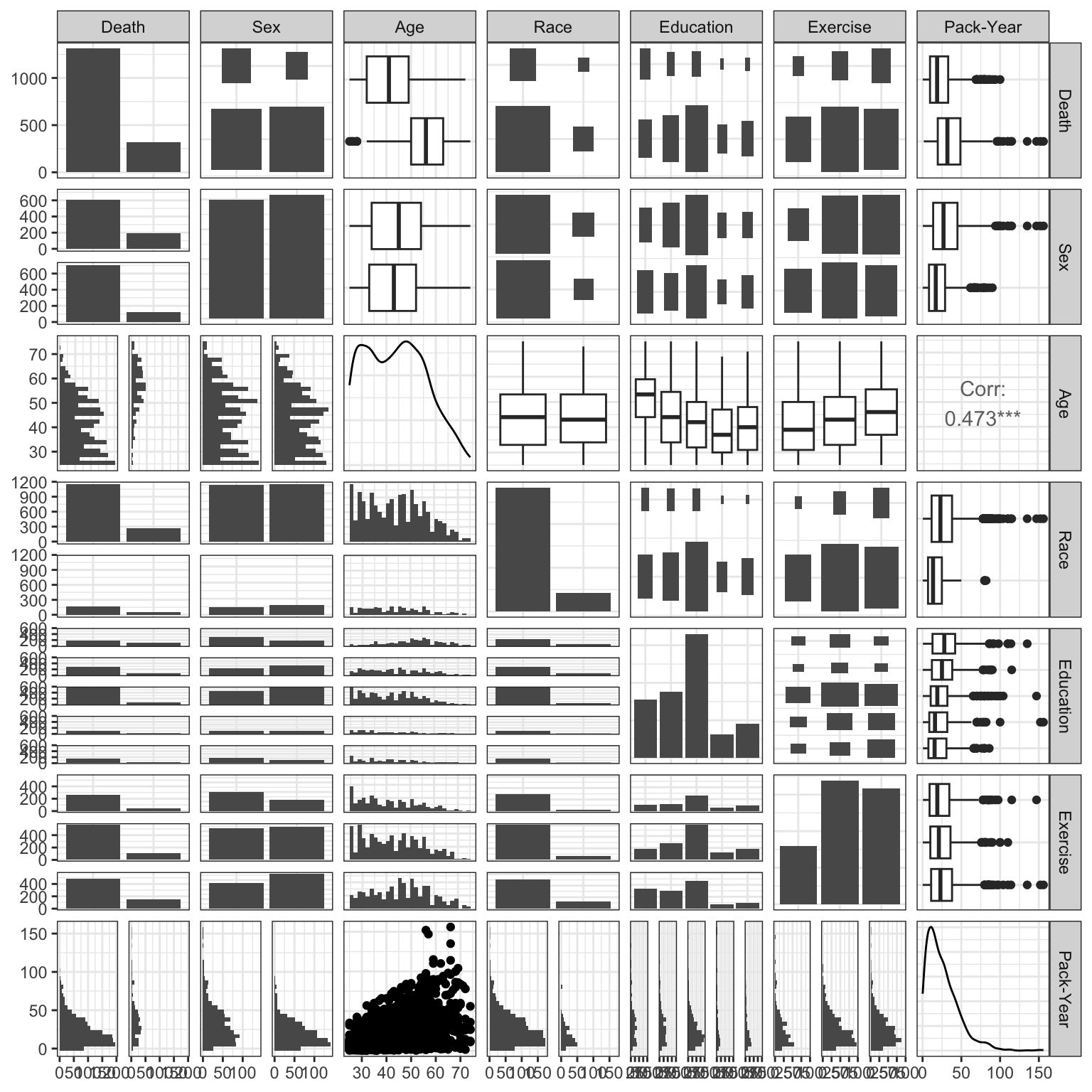
7 【練習1】箱ひげ図+ジッタープロットを描いてみよう!
nhefs データにある心不全の既往歴(hf)と収縮期血圧の変数を使用して、箱ひげ図+ジッタープロットを描いてみましょう。
細かい設定は、下記に示すものとします。それ以外は自由です。
x軸のラベル:Heart failure
y軸のラベル:SBP (mmHg)
Code
ggplot(nhefs04, aes(x = factor(hf), y = sbp, fill = factor(hf))) +
geom_boxplot(outlier.shape = NA) +
geom_jitter(size = 2, alpha = 0.3, width = 0.15, color = "Black") +
scale_fill_manual(values = c("#808080", "#b22222")) +
theme_classic() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 20),
legend.position = "none") +
labs(x = "Heart failure", y = "SBP (mmHg)") +
scale_x_discrete(labels = c("No", "Yes"))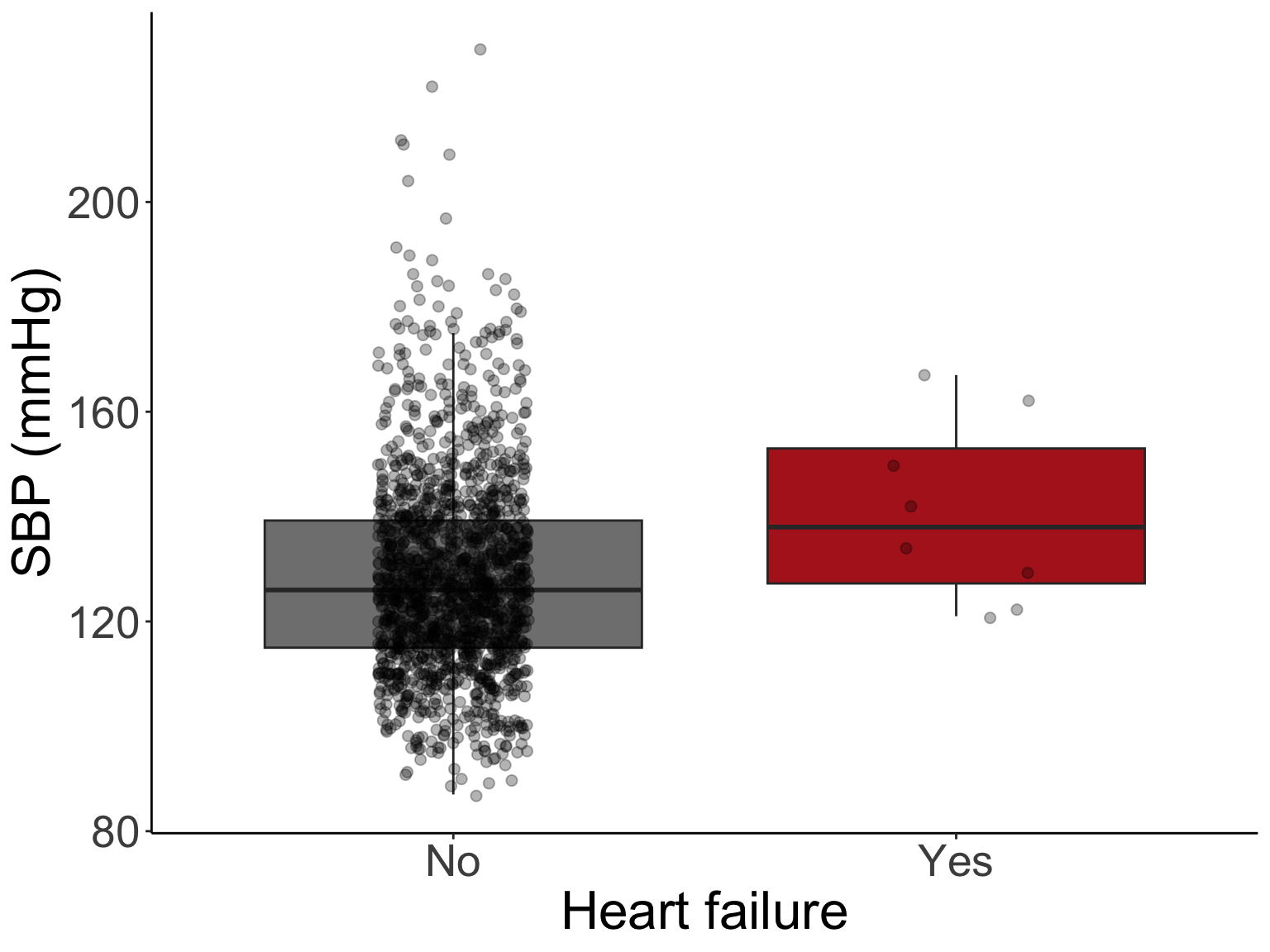
8 【練習2】横並び棒グラフを描いてみよう!
nhefs データにある教育歴と人種の変数を使用して、横並びの棒グラフを描いてみましょう。
細かい設定は、下記に示すものとします。それ以外は自由です。
メインの変数:教育歴
層別化する変数:人種
x軸とy軸を入れ替える
x軸のラベル:Count
y軸のラベル:Education
Code
ggplot(nhefs04, aes(x = education, fill = race)) +
geom_bar(stat = "count", position = "dodge") +
scale_fill_manual(values = c("#2e8b57", "#a52a2a")) +
theme_minimal() +
coord_flip() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 20),
legend.title = element_text(size = 18),
legend.text = element_text(size = 16)) +
labs(x = "Education", y = "Count", fill = "Race") +
guides(fill = guide_legend(reverse = TRUE))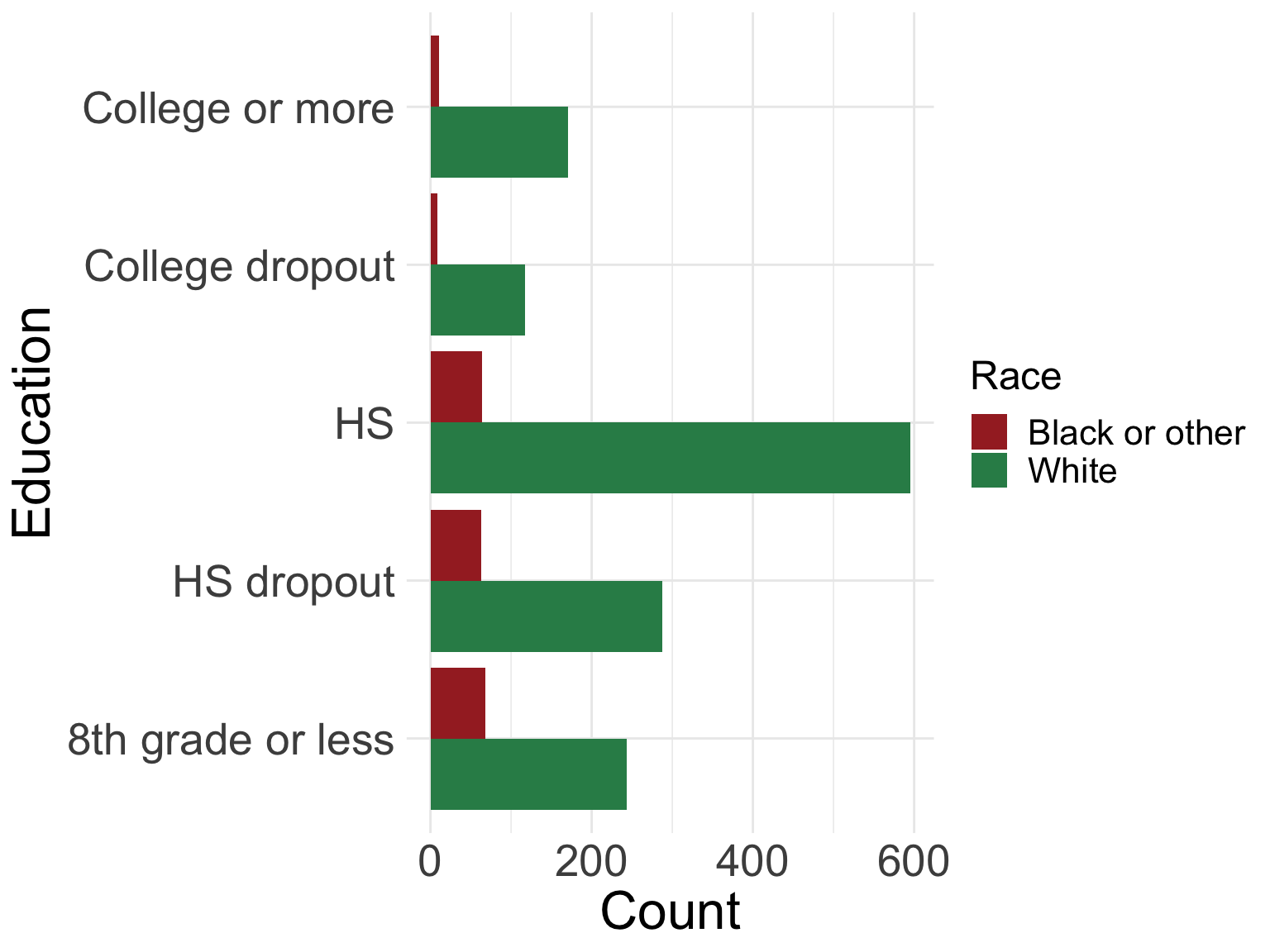
9 【発展】データ加工+ビースウォームプロットを描いてみよう!
Pack-Yearをもとに3つの群(人数は等分になるように)を作り、その3群間で対象者の年齢がどう分布しているかビースウォームプロットを作成しましょう。
三等分した新しい変数を作る時は、cut_number() という関数が便利です。可視化する前に、データを整理してから行いましょう。
今回は何も条件を指定しませんので、これまでの知識を動員して描いてみてください。
Code
# データの加工
nhefs05 <- nhefs04 |>
mutate(
pack_years_n_123 = cut_number(pack_years_n, 3,
labels = c("1st", "2nd", "3rd")))
# 追加した列の確認
# table(nhefs05$pack_years_n_123)
# ビースウォームプロットの描画
ggplot(nhefs05, aes(x = pack_years_n_123, y = age, fill = pack_years_n_123)) +
geom_beeswarm(size = 2, shape = 21, cex = 0.8) +
scale_fill_manual(values = c("#9ECAE1", "#2171B5", "#084594")) +
theme_bw() +
theme(axis.title = element_text(size = 24),
axis.text = element_text(size = 20),
legend.position = "none") +
labs(x = "Pack-Year", y = "Age (years)")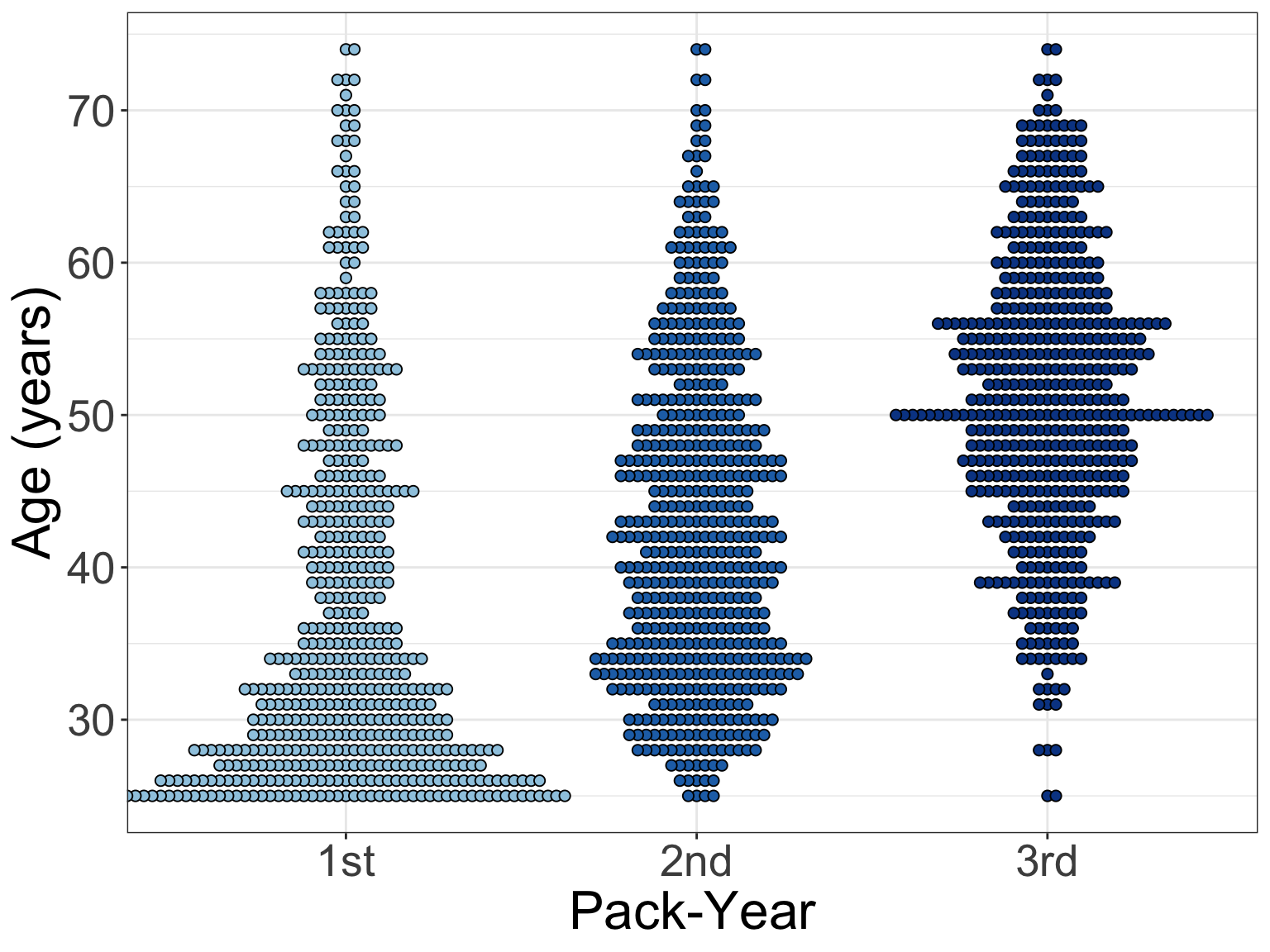
10 まとめ
Part 2では、データを取得した状態から解析前までの可視化を確認しました。データ取得時のミスや各変数の分布を把握することは、その後の解析を進めていく上で欠かせない作業です。
また、記述統計でも、変数ごとに「どのグラフで」「何を」表現するか考えると、非常に奥が深い世界です。より適切なグラフで、簡潔にメッセージを届けるトレーニングを積むことが重要です!
それでは、Part 3でいよいよ生存時間解析です。
Footnotes
Rで使用する主なデータ型には次のようなものがあります。factor:因子型、integer:整数型、numeric:数値型、character:文字列、logical:論理値、NA:欠測↩︎
Warning message が出力されますが、気にしなくても大丈夫です。↩︎
Warning message が出力されますが、気にしなくても大丈夫です。↩︎
Weissgerber TL , et al. Reveal, Don’t Conceal. Transforming Data Visualization to Improve Transparency. Circulation. 2019;140:1506-18.↩︎
Weissgerber TL, et al. Beyond bar and line graphs: time for a new data presentation paradigm. PLoS Biol. 2015;13:e1002128.↩︎
ここで、1や7のカテゴリが抜けていることに気づきます。こうした場合には、「特定のカテゴリが抜けている→データ仕様書等でコーディングを確認する→n=0というのはありえなそうだ→ミスかもしれない」と抜けのないデータクリーニングを行うことができます。↩︎
ここで、1や7のカテゴリが抜けていることに気づきます。こうした場合には、「特定のカテゴリが抜けている→データ仕様書等でコーディングを確認する→n=0というのはありえなそうだ→ミスかもしれない」と抜けのないデータクリーニングを行うことができます。↩︎